num <- 2.2
class(num)[1] "numeric"char <- "hello"
class(char)[1] "character"logi <- TRUE
class(logi)[1] "logical"Jusqu’à présent, vous avez créé des données relativement simples dans R et les avez stockées sous forme de vecteur (Section 2.4). Cependant, la plupart d’entre vous (si ce n’est tous) disposeront d’ensembles de données beaucoup plus complexes provenant de vos diverses expériences et enquêtes, qui vont bien au-delà de ce qu’un vecteur peut gérer. Apprendre comment R traite les différents types de données et structures de données, comment importer vos données dans R et comment manipuler et résumer vos données sont quelques-unes des compétences les plus importantes que vous devrez maîtriser.
Dans ce chapitre, nous passerons en revue les principaux types de données dans R et nous nous concentrerons sur certaines des structures de données les plus courantes. Nous verrons également comment importer des données dans R à partir d’un fichier externe, comment manipuler et résumer des données et enfin comment exporter des données de R vers un fichier externe.
Il est important de comprendre les différents types de données et la manière dont R traite ces données. La tentation est grande d’ignorer ces détails techniques, mais attention, cela peut se retourner contre vous très vite si vous ne faites pas attention. Nous avons déjà vu un exemple (Section 2.3.1) de cela lorsque nous avons essayé (et échoué) d’ajouter deux objets de type caractères ensemble en utilisant l’opérateur +.
R dispose de six types de données de base : numérique (numeric), entier (integer), logique (logical), complexe (complex) et caractère (character). Les plus attentifs d’entre vous remarqueront que nous n’avons listé ici que cinq types de données, le dernier étant le type de données brut (raw), que nous n’aborderons pas car il n’est pas utile dans 99,99 % des cas. Nous n’aborderons pas non plus les nombres complexes, mais nous vous laisserons imaginer cette partie !
Numérique sont des nombres contenant une décimale. En fait, il peut également s’agir de nombres entiers, mais nous n’y reviendrons pas.
Entiers (integer) sont des nombres entiers (sans virgule).
Logique prennent la valeur de TRUE ou FALSE. Il existe également un autre type de logique appelé NA pour représenter les valeurs manquantes.
Caractère sont des chaînes de caractères comme un (ou plusieurs) mot(s). Un type particulier de chaîne de caractères est le facteur qui a des attributs supplémentaires (comme des niveaux ou un ordre). Nous reviendrons sur les facteurs plus tard.
R est (généralement) capable de distinguer automatiquement les différentes classes de données en fonction de leur nature et du contexte dans lequel elles sont utilisées, bien que vous deviez garder à l’esprit que R ne peut pas vraiment lire dans vos pensées et que vous devrez peut-être lui indiquer explicitement comment vous souhaitez traiter un type de données. Vous pouvez connaître le type (ou la classe) de n’importe quel objet en utilisant la fonction class() pour connaître le type (ou la classe) d’un objet.
num <- 2.2
class(num)[1] "numeric"char <- "hello"
class(char)[1] "character"logi <- TRUE
class(logi)[1] "logical"Vous pouvez également demander si un objet appartient à une classe spécifique à l’aide d’un test logique. Le test is.[classOfData]() renvoie soit un TRUE ou un FALSE.
is.numeric(num)[1] TRUEis.character(num)[1] FALSEis.character(char)[1] TRUEis.logical(logi)[1] TRUEIl peut parfois être utile de pouvoir changer la classe d’une variable à l’aide de la fonction as.[className]() bien qu’il faille être prudent car vous pourriez obtenir des résultats inattendus (voir ce qui se passe ci-dessous lorsque nous essayons de convertir une chaîne de caractères en une chaîne numérique).
# convertir un objet numérique en caractère
class(num)[1] "numeric"num_char <- as.character(num)
num_char[1] "2.2"class(num_char)[1] "character"# convertir un objet caractère en numérique !
class(char)[1] "character"char_num <- as.numeric(char)Warning: NAs introduced by coercionVoici un tableau récapitulatif de certaines des fonctions de test logique et de coercion à votre disposition.
| Type de test | Test logique | Coercition |
|---|---|---|
| Caractère | is.character |
as.character |
| Numérique | is.numeric |
as.numeric |
| Logique | is.logical |
as.logical |
| Facteur | is.factor |
as.factor |
| Complexe | is.complex |
as.complex |
Maintenant que vous connaissez certaines des classes de données les plus importantes de R, examinons quelques-unes des principales structures dont nous disposons pour stocker ces données.
Le type de structure de données le plus simple est sans doute le vecteur. Vous avez déjà été initié aux vecteurs dans la Section 2.4, certains des vecteurs que vous avez créés ne contenaient qu’une seule valeur (longueur 1) on. appelle ça un scalaires. Les vecteurs peuvent contenir des nombres, des caractères, des facteurs ou des logiques, mais la chose essentielle à retenir est que tous les éléments à l’intérieur d’un vecteur doivent être de la même classe. En d’autres termes, les vecteurs peuvent contenir des nombres, des caractères ou des logiques, mais pas des mélanges de ces types de données. Il existe une exception importante à cette règle : vous pouvez inclure des NA (rappelez-vous qu’il s’agit d’un type spécial de logique) pour indiquer les données manquantes dans les vecteurs contenant d’autres types de données.
array)La matrice est une autre structure de données utile, utilisée dans de nombreuses disciplines telles que l’écologie des populations et les statistiques théoriques et appliquées. Une matrice est simplement un vecteur doté d’attributs supplémentaires appelés dimensions. Les tableaux (array) ne sont que des matrices multidimensionnelles. Là encore, les matrices et les tableaux doivent contenir des éléments appartenant tous à la même classe de données.
array)
Un moyen pratique de créer une matrice ou un tableau est d’utiliser la fonction matrix() et array() respectivement. Ci-dessous, nous allons créer une matrice à partir d’une séquence de 1 à 16 en quatre lignes (nrow = 4) et la remplir par rangée (byrow = TRUE) plutôt que par colonne, comme c’est le cas par défaut. Lors de l’utilisation de l’option array() il faut définir les dimensions à l’aide de la fonction dim = dans notre cas, 2 lignes, 4 colonnes dans 2 matrices différentes :
ma_mat <- matrix(1:16, nrow = 4, byrow = TRUE)
ma_mat [,1] [,2] [,3] [,4]
[1,] 1 2 3 4
[2,] 5 6 7 8
[3,] 9 10 11 12
[4,] 13 14 15 16, , 1
[,1] [,2] [,3] [,4]
[1,] 1 3 5 7
[2,] 2 4 6 8
, , 2
[,1] [,2] [,3] [,4]
[1,] 9 11 13 15
[2,] 10 12 14 16Il est parfois utile de définir les noms des lignes et des colonnes de votre matrice, mais ce n’est pas obligatoire. Pour ce faire, utilisez la fonction rownames() et colnames() :
a b c d
A 1 2 3 4
B 5 6 7 8
C 9 10 11 12
D 13 14 15 16Une fois que vous avez créé vos matrices, vous pouvez faire des choses utiles avec elles et, comme vous pouvez vous y attendre, R dispose de nombreuses fonctions intégrées pour effectuer des opérations sur les matrices. Certaines des plus courantes sont présentées ci-dessous. Par exemple, pour transposer une matrice, nous utilisons la fonction de transposition t() :
ma_mat_t <- t(ma_mat)
ma_mat_t A B C D
a 1 5 9 13
b 2 6 10 14
c 3 7 11 15
d 4 8 12 16Pour extraire les éléments diagonaux d’une matrice et les stocker sous forme de vecteur, nous pouvons utiliser la fonction diag() :
ma_mat_diag <- diag(ma_mat)
ma_mat_diag[1] 1 6 11 16Les opérations habituelles d’addition, de multiplication, etc. de matrices peuvent être effectuées. Notez l’utilisation de la fonction %*% pour effectuer une multiplication matricielle.
mat.1 <- matrix(c(2, 0, 1, 1), nrow = 2)
mat.1 # Notez que la matrice à été remplie par colonne par défaut [,1] [,2]
[1,] 2 1
[2,] 0 1 [,1] [,2]
[1,] 1 0
[2,] 1 2mat.1 + mat.2 # Addition de matrices [,1] [,2]
[1,] 3 1
[2,] 1 3mat.1 * mat.2 # Produit élément par élément [,1] [,2]
[1,] 2 0
[2,] 0 2mat.1 %*% mat.2 # Multiplication de matrice [,1] [,2]
[1,] 3 2
[2,] 1 2La prochaine structure de données que nous examinerons rapidement est la liste. Alors que les vecteurs et les matrices sont contraints de contenir des données du même type, les listes peuvent stocker des mélanges de types de données. En fait, nous pouvons même stocker d’autres structures de données telles que des vecteurs et des tableaux à l’intérieur d’une liste ou même avoir une liste de liste. Il s’agit donc d’une structure de données très flexible, idéale pour stocker des données irrégulières ou non rectangulaires (voir Chapitre 5 pour un exemple).
Pour créer une liste, nous pouvons utiliser la fonction list(). Notez que les trois éléments de la liste sont de classes différentes (caractère, logique et numérique) et de longueurs différentes.
list_1 <- list(
c("black", "yellow", "orange"),
c(TRUE, TRUE, FALSE, TRUE, FALSE, FALSE),
matrix(1:6, nrow = 3)
)
list_1[[1]]
[1] "black" "yellow" "orange"
[[2]]
[1] TRUE TRUE FALSE TRUE FALSE FALSE
[[3]]
[,1] [,2]
[1,] 1 4
[2,] 2 5
[3,] 3 6Les éléments de la liste peuvent être nommés lors de la construction de la liste :
list_2 <- list(
couleurs = c("black", "yellow", "orange"),
evaluation = c(TRUE, TRUE, FALSE, TRUE, FALSE, FALSE),
temps = matrix(1:6, nrow = 3)
)
list_2$couleurs
[1] "black" "yellow" "orange"
$evaluation
[1] TRUE TRUE FALSE TRUE FALSE FALSE
$temps
[,1] [,2]
[1,] 1 4
[2,] 2 5
[3,] 3 6ou après la création de la liste à l’aide de la fonction names() :
De loin la structure de données la plus couramment utilisée : le jeu de données (data frame). Un jeu de données est un objet bidimensionnel puissant composé de lignes et de colonnes qui ressemble superficiellement à une matrice. Toutefois, alors que les matrices ne peuvent contenir que des données du même type, les jeux de données peuvent contenir un mélange de différents types de données. En règle générale, dans un jeu de données, chaque ligne correspond à une observation individuelle et chaque colonne correspond à une variable mesurée ou enregistrée différente. Cette configuration est peut-être familière à ceux d’entre vous qui utilisent LibreOffice Calc ou Microsoft Excel pour gérer et stocker leurs données. Il peut être utile de penser que les jeux de données sont essentiellement constitués d’un ensemble de vecteurs (colonnes), chaque vecteur contenant son propre type de données, mais le type de données peut être différent d’un vecteur à l’autre.
Par exemple, le jeu de données ci-dessous contient les résultats d’une expérience visant à déterminer l’effet des soins parentaux (avec ou sans) chez les licornes (Unicornus magnificens) sur la croissance des petits sous trois régimes de disponibilité alimentaire différents. Le jeu de données contient 8 variables (colonnes) et chaque ligne représente une licorne individuelle. Les variables p_care et food sont des facteurs (variables catégoriques). La variable p_care a 2 niveaux (care et no_care) et la variable food a 3 niveaux (low, medium et high). Les variables height, weight, mane_size et fluffyness sont numériques et la variable horn_rings est un nombre entier représentant le nombre d’anneaux sur la corne. Bien que la variable block possède des valeurs numériques, celles-ci n’ont pas vraiment d’ordre et pourraient également être traitées comme un facteur (i.e. elles auraient également pu être appelées A et B).
| p_care | food | block | height | weight | mane_size | fluffyness | horn_rings |
|---|---|---|---|---|---|---|---|
| care | medium | 1 | 7.5 | 7.62 | 11.7 | 31.9 | 1 |
| care | medium | 1 | 10.7 | 12.14 | 14.1 | 46.0 | 10 |
| care | medium | 1 | 11.2 | 12.76 | 7.1 | 66.7 | 10 |
| care | medium | 1 | 10.4 | 8.78 | 11.9 | 20.3 | 1 |
| care | medium | 1 | 10.4 | 13.58 | 14.5 | 26.9 | 4 |
| care | medium | 1 | 9.8 | 10.08 | 12.2 | 72.7 | 9 |
| no_care | low | 2 | 3.7 | 8.10 | 10.5 | 60.5 | 6 |
| no_care | low | 2 | 3.2 | 7.45 | 14.1 | 38.1 | 4 |
| no_care | low | 2 | 3.9 | 9.19 | 12.4 | 52.6 | 9 |
| no_care | low | 2 | 3.3 | 8.92 | 11.6 | 55.2 | 6 |
| no_care | low | 2 | 5.5 | 8.44 | 13.5 | 77.6 | 9 |
| no_care | low | 2 | 4.4 | 10.60 | 16.2 | 63.3 | 6 |
Il y a deux choses importantes à garder à l’esprit à propos des jeux de données. Ces types d’objets sont connus sous le nom de données rectangulaires (ou données ordonnées), car chaque colonne doit comporter le même nombre d’observations. Aussi, toute donnée manquante doit être enregistrée sous la forme d’un NA comme nous l’avons fait pour nos vecteurs.
Nous pouvons construire un jeu de données à partir d’objets de données existants, tels que des vecteurs, à l’aide de la fonction data.frame(). À titre d’exemple, créons trois vecteurs p_taille, p_poids et p_noms et incluons tous ces vecteurs dans un jeu de données appelé dataf.
p_taille <- c(180, 155, 160, 167, 181)
p_poids <- c(65, 50, 52, 58, 70)
p_noms <- c("Joanna", "Charlotte", "Helen", "Karen", "Amy")
dataf <- data.frame(taille = p_taille,
poids = p_poids,
noms = p_noms)
dataf taille poids noms
1 180 65 Joanna
2 155 50 Charlotte
3 160 52 Helen
4 167 58 Karen
5 181 70 AmyVous remarquerez que chacune des colonnes est nommée avec le nom de la variable que nous avons fourni lorsque nous avons utilisé la fonction data.frame() lorsque nous avons utilisé la fonction. Il semble également que la première colonne du cadre de données soit une série de nombres allant de 1 à 5. En fait, il ne s’agit pas vraiment d’une colonne, mais du nom de chaque ligne. Nous pouvons le vérifier en demandant à R de renvoyer les dimensions du jeu de données dataf à l’aide de la fonction dim(). Nous constatons qu’il y a 5 lignes et 3 colonnes.
dim(dataf) # 5 lignes et 3 colonnes[1] 5 3Une autre fonction très utile que nous utilisons en permanence est str() qui renvoie un résumé compact de la structure de l’objet data frame (ou de tout autre objet).
str(dataf)'data.frame': 5 obs. of 3 variables:
$ height: num 180 155 160 167 181
$ weight: num 65 50 52 58 70
$ names : chr "Joanna" "Charlotte" "Helen" "Karen" ...La fonction str() nous donne les dimensions du jeu de données et nous rappelle que dataf est un data.frame. Il énumère également toutes les variables (colonnes) contenues dans le jeu de données, nous indique le type de données qu’elles contiennent et leurs 5 premières valeurs. Nous copions souvent ce résumé pour le mettre dans le scripts R avec des commentaires au début de chaque ligne afin de pouvoir nous y référer facilement lors de l’écriture du code. Nous vous avons montré comment commenter les blocs dans RStudio dans la Section 1.7.
Notez également que R a automatiquement décidé que notre p_noms doit être un caractère (chr) lors de la création du jeu de données. La question de savoir si c’est une bonne idée ou non dépendra de la manière dont vous souhaitez utiliser cette variable dans des analyses ultérieures. Si nous décidons que ce n’est pas une bonne idée, nous pouvons modifier le comportement par défaut du data.frame() en incluant l’argument stringsAsFactors = TRUE. Nos chaînes de caractères seront maintenant automatiquement converties en facteurs.
p_taille <- c(180, 155, 160, 167, 181)
p_poids <- c(65, 50, 52, 58, 70)
p_noms <- c("Joanna", "Charlotte", "Helen", "Karen", "Amy")
dataf <- data.frame(
taille = p_taille, poids = p_poids, noms = p_noms,
stringsAsFactors = TRUE
)
str(dataf)'data.frame': 5 obs. of 3 variables:
$ taille: num 180 155 160 167 181
$ poids : num 65 50 52 58 70
$ noms : Factor w/ 5 levels "Amy","Charlotte",..: 4 2 3 5 1Bien que la création de jeux de données à partir de structures de données existantes soit extrêmement utile, l’approche de loin la plus courante consiste à créer un jeu de données en important des données à partir d’un fichier externe. Pour ce faire, vos données doivent être correctement formatées et enregistrées dans un format de fichier que R est capable de reconnaître. Heureusement pour nous, R est capable de reconnaître une grande variété de formats de fichiers, même si, en réalité, vous n’en utiliserez probablement que deux ou trois régulièrement.
La méthode la plus simple pour créer un fichier de données à importer dans R consiste à saisir vos données dans une feuille de calcul à l’aide de Microsoft Excel ou LibreOffice Calc et à l’enregistrer sous la forme d’un fichier délimité par des virgules. Nous préférons LibreOffice Calc car c’est un logiciel libre, indépendant de plate-forme et gratuit, mais MS Excel convient également (mais voir ici pour quelques problèmes). Voici les données de l’expérience sur les licornes dont nous avons parlé précédemment, affichées dans LibreOffice. Si vous souhaitez suivre en même temps que le livre, vous pouvez télécharger le fichier de données (‘unicorn.xlsx’) à partir de l’Annexe A.
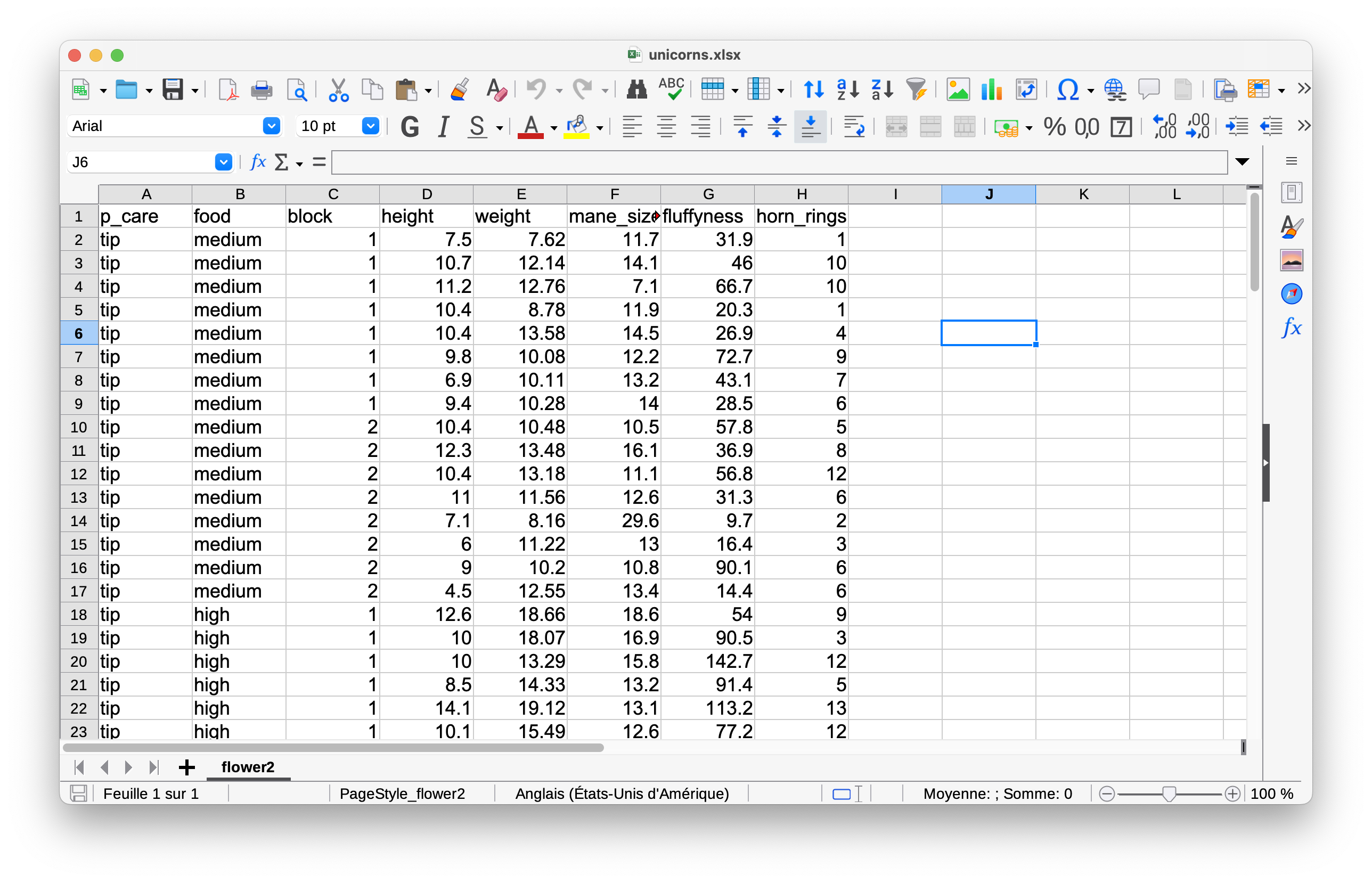
Pour ceux d’entre vous qui ne connaissent pas le format de fichier délimité par des virgules, cela signifie simplement que les données des différentes colonnes sont séparées par le caractère “,” et sont généralement enregistrées dans un fichier portant l’extension “.csv” (Comma-Separated Values).
Pour enregistrer une feuille de calcul en tant que fichier délimité par des virgules (CSV) dans LibreOffice Calc, sélectionnez Fichier -> Enregistrer sous... dans le menu principal. Vous devrez spécifier l’emplacement où vous souhaitez enregistrer votre fichier dans l’option “Enregistrer dans le dossier” et le nom du fichier dans l’option “Nom”. Dans le menu déroulant situé au-dessus du bouton “Enregistrer”, remplacez l’option par défaut “Tous les formats” par “Texte CSV (.csv)”.
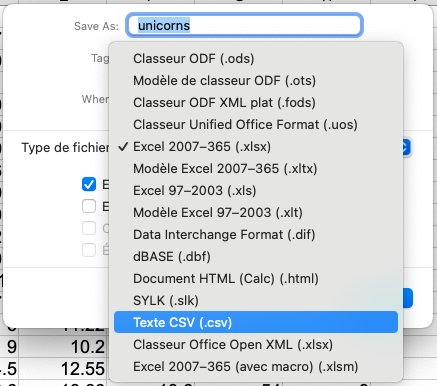
csv lors de l’enregistrement avec LibreOffice Calc
Cliquez sur le bouton Enregistrer, puis sélectionnez l’option “Utiliser le format CSV texte”. Cliquez sur OK pour enregistrer le fichier.
Il y a quelques points à prendre en compte lors de l’enregistrement des fichiers à importer dans R, qui vous faciliteront la vie à long terme. Les titres de vos colonnes (si vous en avez) doivent être courts et informatifs. Évitez également les espaces dans vos titres de colonnes en les remplaçant par un trait de soulignement _ ou un point . (c’est-à-dire remplacer taille de la criniere par taille_de_la_criniere ou taille.de.la.criniere) et d’éviter d’utiliser des caractères spéciaux (par ex. aire (mm^2)), des accents (par ex. écrire criniere au lieu de crinière) ou des majuscules pour vous simplifier la vie. N’oubliez pas que si vous avez des données manquantes dans votre cadre de données (cellules vides), vous devez utiliser un champ NA pour les représenter. Cela permettra de conserver un cadre de données ordonné.
Une fois que vous avez enregistré votre fichier de données dans un format approprié, nous pouvons maintenant lire ce fichier dans R. La fonction la plus utilisée pour importer des données dans R est la fonction read.table() (nous examinerons d’autres solutions plus loin dans ce chapitre). La fonction read.table() est une fonction très flexible qui dispose d’un grand nombre d’arguments (voir ?read.table), mais elle est assez simple à utiliser. Importons le fichier délimité par des virgules appelé unicorns.csv qui contient les données que nous avons vues précédemment dans ce chapitre (Section 3.2.4) que l’on va assigner à un objet appelé licornes. Le fichier est situé dans un dossier data (“données”) qui est lui-même situé dans notre répertoire racine (Section 1.4). La première ligne des données contient les noms des variables (colonnes). Pour utiliser les read.table() pour importer ce fichier
licornes <- read.table(
file = "data/unicorns.csv", header = TRUE, sep = ",", dec = ".",
stringsAsFactors = TRUE
)Il y a quelques points à noter à propos de la commande ci-dessus. Tout d’abord, le chemin d’accès au fichier et le nom du fichier (y compris l’extension) doivent être placés entre guillemets simples ou doubles (c-à-d. le data/unicorns.csv), car la fonction read.table() attend une chaîne de caractères. Si votre répertoire de travail est déjà le répertoire qui contient le fichier, il n’est pas nécessaire d’inclure le chemin d’accès complet au fichier, mais seulement le nom du fichier. Dans l’exemple ci-dessus, le chemin d’accès au fichier est séparé par une simple barre oblique. /. Cela fonctionne quel que soit le système d’exploitation que vous utilisez et nous vous recommandons de vous en tenir à cela. Cependant, les utilisateurs de Windows peuvent être plus familiers avec la notation de la barre oblique inverse simple et si vous voulez continuer à l’utiliser, vous devrez l’inclure en tant que barre oblique inverse double.
Notez cependant que la notation de la double barre oblique inverse fonctionnera pas sur les ordinateurs utilisant les systèmes d’exploitation Mac OSX ou Linux. Nous le déconseillons donc fortement car il n’est pas reproductible
Les header = TRUE spécifie que la première ligne de vos données contient les noms des variables (c.-à-d. food, block etc.) Si ce n’est pas le cas, vous pouvez spécifier header = FALSE (en fait, c’est la valeur par défaut, vous pouvez donc omettre complètement cet argument). L’argument sep = "," indique à R quel est le délimiteur de fichier.
D’autres arguments utiles sont dec = et na.strings =. Les dec = permet de modifier le caractère par défaut (.) utilisé par défaut pour le point décimal. Ceci est utile si vous êtes dans un pays où les décimales sont généralement représentées par une virgule (c.-à-d. dec = ","). L’argument na.strings = vous permet d’importer des données où les valeurs manquantes sont représentées par un symbole autre que NA. Cela peut être assez courant si vous importez des données à partir d’un autre logiciel statistique tel que Minitab, qui représente les valeurs manquantes avec le symbole * (na.strings = "*").
Une série de fonctions prédéfinies sont disponibles dans read.table() pour définire des options spécifiques au format. Mais nous pouvons aussi simplement utiliser read.csv()pour lire un fichier csv, avec la séparation “,” et “.” pour les décimales. Dans les pays où “,” est utilisé pour les décimales, les fichiers csv utilisent “;” comme séparateur. Dans ce cas, l’utilisation de read.csv2() serait nécessaire. Lorsque l’on travaille avec des fichiers délimités par des tabulations, les fonctions read.delim() et read.delim2() peuvent être utilisées avec “.” et “,” comme décimales respectivement.
Après avoir importé nos données dans R, pour voir le contenu du jeu de données, il suffit de taper le nom de l’objet comme nous l’avons fait précédemment. MAIS avant de faire cela, réfléchissez à la raison pour laquelle vous faites cela. Si votre jeu de données est autre chose que minuscule, tout ce que vous allez faire, c’est remplir votre Console de données. Ce n’est pas comme si vous pouviez facilement vérifier s’il y a des erreurs ou si vos données ont été importées correctement. Une bien meilleure solution consiste à utiliser notre vieil ami, la fonction str() pour obtenir un résumé compact et informatif de votre base de données.
str(licornes)'data.frame': 96 obs. of 8 variables:
$ p_care : Factor w/ 2 levels "care","no_care": 1 1 1 1 1 1 1 1 1 1 ...
$ food : Factor w/ 3 levels "high","low","medium": 3 3 3 3 3 3 3 3 3 3 ...
$ block : int 1 1 1 1 1 1 1 1 2 2 ...
$ height : num 7.5 10.7 11.2 10.4 10.4 9.8 6.9 9.4 10.4 12.3 ...
$ weight : num 7.62 12.14 12.76 8.78 13.58 ...
$ mane_size : num 11.7 14.1 7.1 11.9 14.5 12.2 13.2 14 10.5 16.1 ...
$ fluffyness: num 31.9 46 66.7 20.3 26.9 72.7 43.1 28.5 57.8 36.9 ...
$ horn_rings: int 1 10 10 1 4 9 7 6 5 8 ...Ici, nous voyons que licornes est un objet “data.frame” qui contient 96 lignes et 8 variables (colonnes). Chacune des variables est répertoriée avec sa classe de données et les 10 premières valeurs. Comme nous l’avons mentionné précédemment dans ce chapitre, il peut être très pratique de copier/coller ces données dans votre script R sous la forme d’un bloc de commentaires afin de pouvoir s’y référer ultérieurement.
Notez également que vos variables de type chaîne de caractères (care et food) ont été importées en tant que facteurs parce que nous avons utilisé l’argument stringsAsFactors = TRUE. Si ce n’est pas ce que vous voulez, vous pouvez utiliser l’argument stringsAsFactors = FALSE ou à partir de la version 4.0.0 de R, vous pouvez simplement ne pas utiliser cet argument car stringsAsFactors = FALSE est la valeur par défaut.
Nous pouvons donc aussi extraire le jeu de données depuis un fichier texte (.txt) grâce à la fonction read.delim() :
licornes <- read.delim(file = "data/unicorns.txt")
str(licornes)'data.frame': 96 obs. of 8 variables:
$ p_care : chr "care" "care" "care" "care" ...
$ food : chr "medium" "medium" "medium" "medium" ...
$ block : int 1 1 1 1 1 1 1 1 2 2 ...
$ height : num 7.5 10.7 11.2 10.4 10.4 9.8 6.9 9.4 10.4 12.3 ...
$ weight : num 7.62 12.14 12.76 8.78 13.58 ...
$ mane_size : num 11.7 14.1 7.1 11.9 14.5 12.2 13.2 14 10.5 16.1 ...
$ fluffyness: num 31.9 46 66.7 20.3 26.9 72.7 43.1 28.5 57.8 36.9 ...
$ horn_rings: int 1 10 10 1 4 9 7 6 5 8 ...Si nous voulons simplement voir les noms de nos variables (colonnes) dans le jeu de données, nous pouvons utiliser l’argument names() qui renverra un vecteur de caractères contenant les noms des variables.
names(licornes)[1] "p_care" "food" "block" "height" "weight"
[6] "mane_size" "fluffyness" "horn_rings"Vous pouvez même importer des feuilles de calcul de MS Excel ou d’autres logiciels de statistiques directement dans R, mais nous vous conseillons d’éviter cette méthode dans la mesure du possible, car elle ne fait qu’ajouter une couche d’incertitude entre vous et vos données. À notre avis, il est presque toujours préférable d’exporter vos feuilles de calcul sous forme de fichiers délimités par des tabulations (.txt) ou des virgules (.csv), puis de les importer dans R à l’aide de l’un des fonctions dérivées de read.table() (c-à-d. read.delim(), read.csv(), etc.). Si vous tenez absolument à importer directement des données à partir d’un autre logiciel, vous devrez installer le paquet foreign 📦 qui contient des fonctions permettant d’importer des fichiers Minitab, SPSS, Stata et SAS. Pour les feuilles de calcul MS Excel et LO Calc, quelques paquets peuvent être utilisés.
Il est assez courant d’obtenir un tas de messages d’erreur vraiment frustrants lorsque l’on commence à importer des données dans R. Le plus courant est sans doute
Error in file(file, "rt") : cannot open the connection
In addition: Warning message:
In file(file, "rt") :
cannot open file 'unicorns.txt': No such file or directoryCe message d’erreur vous indique que R ne peut pas trouver le fichier que vous essayez d’importer. Il apparaît généralement pour l’une ou l’autre des raisons suivantes (ou pour toutes !). La première est que vous avez fait une erreur dans l’orthographe du nom de fichier ou du chemin d’accès au fichier. Une autre erreur fréquente est d’avoir oublié d’inclure l’extension du fichier dans le nom du fichier (par ex. .txt). Enfin, le fichier ne se trouve pas à l’endroit indiqué ou vous avez utilisé un chemin d’accès incorrect. L’utilisation de projets RStudio (Section 1.5) et d’une structure de répertoire logique (Section 1.4) permet d’éviter (limiter) ce type d’erreurs.
Une autre erreur très fréquente est d’oublier d’inclure l’élément header = TRUE lorsque la première ligne des données contient des noms de variables. Par exemple, si nous omettons cet argument lorsque nous importons notre fichier unicorns.txt tout semble correct au début (pas de message d’erreur au moins).
licornes_erreur <- read.table(file = "data/unicorns.txt", sep = "\t")mais lorsque nous jetons un coup d’œil à notre jeu de données en utilisant str()
str(licornes_erreur)'data.frame': 97 obs. of 8 variables:
$ V1: chr "p_care" "care" "care" "care" ...
$ V2: chr "food" "medium" "medium" "medium" ...
$ V3: chr "block" "1" "1" "1" ...
$ V4: chr "height" "7.5" "10.7" "11.2" ...
$ V5: chr "weight" "7.62" "12.14" "12.76" ...
$ V6: chr "mane_size" "11.7" "14.1" "7.1" ...
$ V7: chr "fluffyness" "31.9" "46" "66.7" ...
$ V8: chr "horn_rings" "1" "10" "10" ...Nous constatons un problème évident, toutes nos variables ont été importées en tant que facteurs et sont nommées V1, V2, V3 … V8. Le problème vient du fait que nous n’avons pas dit au read.table() que la première ligne contient les noms des variables et donc elle les traite comme des données. Dès que nous avons une chaîne de caractères dans l’une de nos variable, R les traite comme des données de type caractère (rappelez-vous que tous les éléments d’un vecteur doivent contenir le même type de données (Section 3.2.1)).
Ce n’est qu’un argument de plus pour utiliser read.csv() ou read.delim() avec des valeurs par défaut appropriées pour les arguments.
Il existe de nombreuses autres fonctions permettant d’importer des données à partir d’une variété de sources et de formats. La plupart de ces fonctions sont contenues dans des paquets que vous devez installer avant de les utiliser. Vous trouverez ci-dessous une liste des paquets et des fonctions les plus utiles.
Les paquets fread() du paquet data.table 📦 est idéale pour importer rapidement et efficacement de grands fichiers de données (beaucoup plus rapidement que la fonction read.table()). L’un des aspects les plus intéressants de la fonction fread() est qu’elle détecte automatiquement la plupart des arguments que vous devriez normalement spécifier (comme sep = etc.) Une choses à prendre en compte cependant est que la fonction fread() renverra un objet de type data.table et non data.frame comme ce serait le cas avec la fonction read.table(). Ce n’est généralement pas un problème, car vous pouvez passer un objet data.table à n’importe quelle fonction n’acceptant normalement que des objets data.frame. Pour en savoir plus sur les différences entre data.table et data.frame voir ici .
library(data.table)
toutes_donnees <- fread(file = "data/unicorns.txt")Diverses fonctions du paquet readr 📦 sont également très efficaces pour lire des fichiers de données volumineux. Le paquet readr 📦 fait partie de la collection de paquet ‘tidyverse’ et fournit de nombreuses fonctions équivalentes à celles de la version de base de R pour l’importation de données. Les fonctions de readr sont utilisées de la même manière que les fonctions read.table() ou read.csv() et la plupart des arguments sont les mêmes (voir ?readr::read_table pour plus de détails). Il existe cependant quelques différences. Par exemple, lors de l’utilisation de l’option read_table() l’argument header = TRUE est remplacé par col_names = TRUE et la fonction renvoie un objet de classe tibble qui est l’équivalent dans le tidyverse d’un objet de classe data.frame (voir ici pour les différences).
Attention ! certain.e.s de vos modèles ou fonctions, etc. peuvent ne pas fonctionner ou fonctionner différement à cause de ça (vérifier/reconvertir votre jeu de données au format data.frame peut vous économiser du temps de débeugage).
library(readr)
# importation de fichiers délimités par des espaces blancs
toutes_donnees <- read_table(file = "data/unicorns.txt", col_names = TRUE)
# importation de fichiers délimités par des virgules
toutes_donnees <- read_csv(file = "data/unicorns.csv")
# importation de fichiers délimités par des tabulations
toutes_donnees <- read_delim(file = "data/unicorns.txt", delim = "\t")
# ou utilisez
toutes_donnees <- read_tsv(file = "data/unicorns.txt")Si votre fichier de données est énorme, les paquets ff 📦 et bigmemory 📦 peuvent être utiles car ils contiennent tous deux des fonctions d’importation capables de stocker des données volumineuses de manière efficace en termes de mémoire. Pour en savoir plus sur ces fonctions ici et ici .
Maintenant que vous avez réussi à importer vos données dans R depuis un fichier externe, la prochaine étape est de faire quelque chose d’utile avec nos données. La manipulation de données est une compétence fondamentale que vous devrez développer et avec laquelle vous devrez vous sentir à l’aise, car vous en ferez probablement beaucoup au cours de n’importe quel projet. La bonne nouvelle, c’est que R est particulièrement efficace pour manipuler, résumer et visualiser les données. La manipulation de données (souvent connue sous le nom de “data wrangling” ou “munging”) en R peut sembler un peu intimidante au début pour un nouvel utilisateur, mais si vous suivez quelques règles logiques simples, vous prendrez rapidement le coup de main, surtout avec un peu de pratique.
Rappelons la structure du jeu de données licornes que nous avons importée dans la section précédente :
licornes <- read.table(file = "data/unicorns.txt", header = TRUE, sep = "\t")
str(licornes)'data.frame': 96 obs. of 8 variables:
$ p_care : chr "care" "care" "care" "care" ...
$ food : chr "medium" "medium" "medium" "medium" ...
$ block : int 1 1 1 1 1 1 1 1 2 2 ...
$ height : num 7.5 10.7 11.2 10.4 10.4 9.8 6.9 9.4 10.4 12.3 ...
$ weight : num 7.62 12.14 12.76 8.78 13.58 ...
$ mane_size : num 11.7 14.1 7.1 11.9 14.5 12.2 13.2 14 10.5 16.1 ...
$ fluffyness: num 31.9 46 66.7 20.3 26.9 72.7 43.1 28.5 57.8 36.9 ...
$ horn_rings: int 1 10 10 1 4 9 7 6 5 8 ...Pour accéder aux données de n’importe quelle variable (colonne) de notre jeu de données, nous pouvons utiliser la notation $. Par exemple, pour accéder à la variable height dans le jeu de données licornes on écrit licornes$height. Cela indique à R que la variable height est contenue dans le jeu de données licornes.
licornes$height [1] 7.5 10.7 11.2 10.4 10.4 9.8 6.9 9.4 10.4 12.3 10.4 11.0 7.1 6.0 9.0
[16] 4.5 12.6 10.0 10.0 8.5 14.1 10.1 8.5 6.5 11.5 7.7 6.4 8.8 9.2 6.2
[31] 6.3 17.2 8.0 8.0 6.4 7.6 9.7 12.3 9.1 8.9 7.4 3.1 7.9 8.8 8.5
[46] 5.6 11.5 5.8 5.6 5.3 7.5 4.1 3.5 8.5 4.9 2.5 5.4 3.9 5.8 4.5
[61] 8.0 1.8 2.2 3.9 8.5 8.5 6.4 1.2 2.6 10.9 7.2 2.1 4.7 5.0 6.5
[76] 2.6 6.0 9.3 4.6 5.2 3.9 2.3 5.2 2.2 4.5 1.8 3.0 3.7 2.4 5.7
[91] 3.7 3.2 3.9 3.3 5.5 4.4Cette opération renvoie un vecteur des données de la variable height. Si nous le souhaitons, nous pouvons assigner ce vecteur à un autre objet et faire d’autres choses avec, comme calculer une moyenne ou obtenir un résumé de la variable à l’aide de la fonction summary() :
f_taille <- licornes$height
mean(f_taille)[1] 6.839583summary(f_taille) Min. 1st Qu. Median Mean 3rd Qu. Max.
1.200 4.475 6.450 6.840 9.025 17.200 Ou si nous ne voulons pas créer un objet supplémentaire, nous pouvons utiliser des fonctions “à la volée” pour afficher uniquement la valeur dans la console.
mean(licornes$height)[1] 6.839583summary(licornes$height) Min. 1st Qu. Median Mean 3rd Qu. Max.
1.200 4.475 6.450 6.840 9.025 17.200 Tout comme nous l’avons fait avec les vecteurs (Section 2.5), nous pouvons également accéder aux données contenues dans le jeu de données en utilisant les crochets [ ]. Cependant, au lieu d’utiliser un seul index, nous devons maintenant utiliser deux index, l’un pour spécifier les lignes et l’autre pour les colonnes. Pour ce faire, nous pouvons utiliser la notation mes_donnees[ligne, colonne] où ligne et colonne sont des indices et mes_donnees est le nom du jeu de données. Une fois encore, comme pour nos vecteurs, nos index peuvent être positionnels ou résulter d’un test logique.
Pour utiliser les index positionnels, il suffit d’écrire la position des lignes et des colonnes que l’on veut extraire à l’intérieur des crochets [ ]. Par exemple, si, pour une raison quelconque, nous voulons extraire la première valeur (1ère ligne) de la variable height (4e colonne) :
licornes[1, 4][1] 7.5# on peut obtenir le même résultat avec cette notation :
licornes$height[1][1] 7.5Nous pouvons également extraire les valeurs de plusieurs lignes ou colonnes en spécifiant ces index sous forme de vecteurs à l’intérieur des crochets [ ]. Pour extraire les 10 premières lignes et les 4 premières colonnes, il suffit de fournir un vecteur contenant une séquence de 1 à 10 pour l’index des lignes (1:10) et un vecteur de 1 à 4 pour l’index des colonnes (1:4) :
licornes[1:10, 1:4] p_care food block height
1 care medium 1 7.5
2 care medium 1 10.7
3 care medium 1 11.2
4 care medium 1 10.4
5 care medium 1 10.4
6 care medium 1 9.8
7 care medium 1 6.9
8 care medium 1 9.4
9 care medium 2 10.4
10 care medium 2 12.3Si les lignes et les colonnes ne sont pas séquentielles, nous pouvons fournir des vecteurs de positions à l’aide de la fonction c(). Pour extraire les 1ère, 5e, 12e et 30e lignes des 1ère, 3e, 6e et 8e colonnes :
p_care block mane_size horn_rings
1 care 1 11.7 1
5 care 1 14.5 4
12 care 2 12.6 6
30 care 2 11.6 5Tout ce que nous faisons dans les deux exemples ci-dessus est de créer des vecteurs de positions pour les lignes et les colonnes que nous voulons extraire. Pour ce faire, nous avons utilisé les compétences que nous avons développées dans la Section 2.4 lorsque nous avons généré des vecteurs à l’aide de la fonction c() ou en utilisant la notation :.
Mais qu’en est-il si nous voulons extraire toutes les lignes ou toutes les colonnes ? Il serait extrêmement fastidieux de devoir générer des vecteurs pour toutes les lignes ou pour toutes les colonnes. Heureusement, R dispose d’un raccourci. Si vous ne spécifiez pas d’index de ligne ou de colonne dans les crochets [ ] R interprète cela comme signifiant que vous voulez toutes les lignes ou toutes les colonnes. Par exemple, pour extraire les 4 premières lignes et toutes les colonnes du jeu de données licornes :
licornes[1:4, ] p_care food block height weight mane_size fluffyness horn_rings
1 care medium 1 7.5 7.62 11.7 31.9 1
2 care medium 1 10.7 12.14 14.1 46.0 10
3 care medium 1 11.2 12.76 7.1 66.7 10
4 care medium 1 10.4 8.78 11.9 20.3 1ou toutes les lignes et les 3 premières colonnes 1 .
unicorns[, 1:3] p_care food block
1 care medium 1
2 care medium 1
3 care medium 1
4 care medium 1
5 care medium 1
92 no_care low 2
93 no_care low 2
94 no_care low 2
95 no_care low 2
96 no_care low 2Nous pouvons même utiliser des index de position négatifs pour exclure certaines lignes et colonnes. Par exemple, extrayons toutes les lignes à l’exception des 85 premières et toutes les colonnes à l’exception de la 4e, 7e et 8e. Remarquez que nous devons utiliser -() lorsque nous générons nos vecteurs de position de ligne. Si nous avions simplement utilisé -1:85 cela générerait en fait une séquence régulière de -1 à 85, ce qui n’est pas ce que nous voulons (nous pouvons bien sûr utiliser -1:-85).
licornes[-(1:85), -c(4, 7, 8)] p_care food block weight mane_size
86 no_care low 1 6.01 17.6
87 no_care low 1 9.93 12.0
88 no_care low 1 7.03 7.9
89 no_care low 2 9.10 14.5
90 no_care low 2 9.05 9.6
91 no_care low 2 8.10 10.5
92 no_care low 2 7.45 14.1
93 no_care low 2 9.19 12.4
94 no_care low 2 8.92 11.6
95 no_care low 2 8.44 13.5
96 no_care low 2 10.60 16.2Outre l’utilisation d’un index de position pour extraire des colonnes (variables) particulières, nous pouvons également nommer les variables directement en utilisant les crochet [ ]. Par exemple, extrayons les 5 premières lignes des variables care, food et mane_size. Au lieu d’utiliser licornes[1:5, c(1, 2, 6)] nous pouvons utiliser :
licornes[1:5, c("p_care", "food", "mane_size")] p_care food mane_size
1 care medium 11.7
2 care medium 14.1
3 care medium 7.1
4 care medium 11.9
5 care medium 14.5En général, on préférera utiliser cette méthode plutôt que l’index positionnel pour sélectionner les colonnes, car elle nous donnera toujours ce que nous voulons même si nous avons changé l’ordre des colonnes dans notre jeu de données pour une raison quelconque.
Tout comme nous l’avons fait avec les vecteurs, nous pouvons également extraire des données de notre jeu de données sur la base d’un test logique. Nous pouvons utiliser tous les opérateurs logiques que nous avons utilisés dans les exemples des vecteurs. Si ceux-ci vous ont échappé, jetez un coup d’œil à la Section 2.5.1.1 pour vous rafraîchir la mémoire. Extrayons toutes les lignes pour lesquelles la variable height a une valeur supérieure à 12 et extrayons toutes les colonnes par défaut (rappelez-vous, si vous n’incluez pas d’index de colonne après la virgule, cela signifie toutes les colonnes).
grandes_cornes <- licornes[licornes$height > 12, ]
grandes_cornes p_care food block height weight mane_size fluffyness horn_rings
10 care medium 2 12.3 13.48 16.1 36.9 8
17 care high 1 12.6 18.66 18.6 54.0 9
21 care high 1 14.1 19.12 13.1 113.2 13
32 care high 2 17.2 19.20 10.9 89.9 14
38 care low 1 12.3 11.27 13.7 28.7 5Remarquez dans le code ci-dessus qu’il faut utiliser l’élément licornes$height pour le test logique. Si nous nommions simplement la variable height sans le nom du jeu de données, nous recevrions une erreur nous indiquant que R ne peut pas trouver la variable height. La raison en est que la variable height n’existe qu’à l’intérieur du jeu de données licornes vous devez donc indiquer à R où elle se trouve exactement.
grandes_cornes <- licornes[height > 12, ]
Error in `[.data.frame`(licornes, height > 12, ) :
object 'height' not foundComment cela fonctionne-t-il ? Le test logique est le suivant licornes$height > 12 et R n’extraira que les lignes qui satisfont à cette condition logique. Si nous regardons la sortie de la seule condition logique, vous pouvez voir qu’elle renvoie un vecteur contenant TRUE si height est supérieur à 12 et FALSE si height n’est pas supérieur à 12.
licornes$height > 12 [1] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE TRUE FALSE FALSE
[13] FALSE FALSE FALSE FALSE TRUE FALSE FALSE FALSE TRUE FALSE FALSE FALSE
[25] FALSE FALSE FALSE FALSE FALSE FALSE FALSE TRUE FALSE FALSE FALSE FALSE
[37] FALSE TRUE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[49] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[61] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[73] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[85] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSENotre indice de ligne est donc un vecteur contenant soit TRUE ou FALSE et seulement les lignes qui sont TRUE sont sélectionnées.
D’autres opérateurs couramment utilisés sont présentés ci-dessous :
licornes[licornes$height >= 6, ] # valeurs supérieures ou égales à 6
licornes[licornes$height <= 6, ] # valeurs inférieures ou égales à 6
licornes[licornes$height == 8, ] # valeurs égales à 8
licornes[licornes$height != 8, ] # valeurs différentes de 8Nous pouvons également extraire des lignes en fonction de la valeur d’une chaîne de caractères ou d’un niveau de facteur. Extrayons toutes les lignes pour lesquelles la valeur de la variable food est égal à high (là encore, nous extrairons toutes les colonnes). Remarquez que la double égalité == doit être utilisé pour un test logique et que la chaîne de caractères doit être placée entre guillemets simples ou doubles (c.-à-d. "high").
nourriture_bcp <- licornes[licornes$food == "high", ]
rbind(head(nourriture_bcp, n = 10), tail(nourriture_bcp, n = 10)) p_care food block height weight mane_size fluffyness horn_rings
17 care high 1 12.6 18.66 18.6 54.0 9
18 care high 1 10.0 18.07 16.9 90.5 3
19 care high 1 10.0 13.29 15.8 142.7 12
20 care high 1 8.5 14.33 13.2 91.4 5
21 care high 1 14.1 19.12 13.1 113.2 13
22 care high 1 10.1 15.49 12.6 77.2 12
23 care high 1 8.5 17.82 20.5 54.4 3
24 care high 1 6.5 17.13 24.1 147.4 6
25 care high 2 11.5 23.89 14.3 101.5 12
26 care high 2 7.7 14.77 17.2 104.5 4
71 no_care high 1 7.2 15.21 15.9 135.0 14
72 no_care high 1 2.1 19.15 15.6 176.7 6
73 no_care high 2 4.7 13.42 19.8 124.7 5
74 no_care high 2 5.0 16.82 17.3 182.5 15
75 no_care high 2 6.5 14.00 10.1 126.5 7
76 no_care high 2 2.6 18.88 16.4 181.5 14
77 no_care high 2 6.0 13.68 16.2 133.7 2
78 no_care high 2 9.3 18.75 18.4 181.1 16
79 no_care high 2 4.6 14.65 16.7 91.7 11
80 no_care high 2 5.2 17.70 19.1 181.1 8Ou nous pouvons extraire toutes les lignes où food est différent de medium (en utilisant !=) et ne sélectionner que les colonnes 1 à 4.
nourriture_pas_moyenne <- licornes[licornes$food != "medium", 1:4]
rbind(head(nourriture_pas_moyenne, n = 10), tail(nourriture_pas_moyenne, n = 10)) p_care food block height
17 care high 1 12.6
18 care high 1 10.0
19 care high 1 10.0
20 care high 1 8.5
21 care high 1 14.1
22 care high 1 10.1
23 care high 1 8.5
24 care high 1 6.5
25 care high 2 11.5
26 care high 2 7.7
87 no_care low 1 3.0
88 no_care low 1 3.7
89 no_care low 2 2.4
90 no_care low 2 5.7
91 no_care low 2 3.7
92 no_care low 2 3.2
93 no_care low 2 3.9
94 no_care low 2 3.3
95 no_care low 2 5.5
96 no_care low 2 4.4Nous pouvons accroître la complexité de nos tests logiques en les combinant avec des des expressions booléennes comme nous l’avons fait pour les objets vectoriels. Par exemple, pour extraire toutes les lignes où la taille (height) est supérieure ou égale à 6 ET food est égal à medium ET care est égal à no_care nous combinons une série d’expressions logiques avec le symbole & :
faible_soins_non_taille6 <- licornes[licornes$height >= 6 &
licornes$food == "medium" &
licornes$p_care == "no_care", ]
faible_soins_non_taille6 p_care food block height weight mane_size fluffyness horn_rings
51 no_care medium 1 7.5 13.60 13.6 122.2 11
54 no_care medium 1 8.5 10.04 12.3 113.6 4
61 no_care medium 2 8.0 11.43 12.6 43.2 14Pour extraire des lignes sur la base d’une expression booléenne “OR” (“OU”), nous pouvons utiliser le symbole |. Extrayons toutes les lignes où la taille (height) est supérieure à 12,3 OU inférieure à 2,2.
taille2.2_12.3 <- licornes[licornes$height > 12.3 | licornes$height < 2.2, ]
taille2.2_12.3 p_care food block height weight mane_size fluffyness horn_rings
17 care high 1 12.6 18.66 18.6 54.0 9
21 care high 1 14.1 19.12 13.1 113.2 13
32 care high 2 17.2 19.20 10.9 89.9 14
62 no_care medium 2 1.8 10.47 11.8 120.8 9
68 no_care high 1 1.2 18.24 16.6 148.1 7
72 no_care high 1 2.1 19.15 15.6 176.7 6
86 no_care low 1 1.8 6.01 17.6 46.2 4Une autre méthode pour sélectionner des parties d’un jeu de données sur la base d’une expression logique consiste à utiliser la fonction subset() au lieu des crochets [ ]. L’avantage d’utiliser subset() est qu’il n’est plus nécessaire d’utiliser le symbole $ pour spécifier des variables à l’intérieur du jeu de données, car le premier argument de la fonction est le nom du jeu de données à subdiviser. L’inconvénient est que subset() est moins flexible que la notation [ ].
soins_moy_2 <- subset(licornes, p_care == "care" & food == "medium" & block == 2)
soins_moy_2 p_care food block height weight mane_size fluffyness horn_rings
9 care medium 2 10.4 10.48 10.5 57.8 5
10 care medium 2 12.3 13.48 16.1 36.9 8
11 care medium 2 10.4 13.18 11.1 56.8 12
12 care medium 2 11.0 11.56 12.6 31.3 6
13 care medium 2 7.1 8.16 29.6 9.7 2
14 care medium 2 6.0 11.22 13.0 16.4 3
15 care medium 2 9.0 10.20 10.8 90.1 6
16 care medium 2 4.5 12.55 13.4 14.4 6Et si vous ne voulez que certaines colonnes, vous pouvez utiliser l’arguement select = :
uni_p_care <- subset(licornes, p_care == "care" & food == "medium" & block == 2,
select = c("p_care", "food", "mane_size")
)
uni_p_care p_care food mane_size
9 care medium 10.5
10 care medium 16.1
11 care medium 11.1
12 care medium 12.6
13 care medium 29.6
14 care medium 13.0
15 care medium 10.8
16 care medium 13.4Rappelez-vous lorsque nous avons utilisé la fonction order() pour ordonner un vecteur en fonction de l’ordre d’un autre vecteur (dans la Section 2.5.3). Cette fonction est très utile si vous souhaitez réorganiser les lignes de votre jeu de données. Par exemple, si nous voulons que toutes les lignes du jeu de données licornes soient classées par ordre croissant en fonction de la variable height et éditer toutes les colonnes par défaut :
p_care food block height weight mane_size fluffyness horn_rings
68 no_care high 1 1.2 18.24 16.6 148.1 7
62 no_care medium 2 1.8 10.47 11.8 120.8 9
86 no_care low 1 1.8 6.01 17.6 46.2 4
72 no_care high 1 2.1 19.15 15.6 176.7 6
63 no_care medium 2 2.2 10.70 15.3 97.1 7
84 no_care low 1 2.2 9.97 9.6 63.1 2
82 no_care low 1 2.3 7.28 13.8 32.8 6
89 no_care low 2 2.4 9.10 14.5 78.7 8
56 no_care medium 1 2.5 14.85 17.5 77.8 10
69 no_care high 1 2.6 16.57 17.1 141.1 3Nous pouvons également ordonner par ordre décroissant d’une variable (i.e. mane_size) en utilisant l’argument decreasing = TRUE :
taille_criniere_ord <- licornes[order(licornes$mane_size, decreasing = TRUE), ]
head(taille_criniere_ord, n = 10) p_care food block height weight mane_size fluffyness horn_rings
70 no_care high 1 10.9 17.22 49.2 189.6 17
13 care medium 2 7.1 8.16 29.6 9.7 2
24 care high 1 6.5 17.13 24.1 147.4 6
65 no_care high 1 8.5 22.53 20.8 166.9 16
23 care high 1 8.5 17.82 20.5 54.4 3
66 no_care high 1 8.5 17.33 19.8 184.4 12
73 no_care high 2 4.7 13.42 19.8 124.7 5
80 no_care high 2 5.2 17.70 19.1 181.1 8
17 care high 1 12.6 18.66 18.6 54.0 9
49 no_care medium 1 5.6 11.03 18.6 49.9 8Nous pouvons même ordonner des jeux de données sur la base de plusieurs variables. Par exemple, pour ordonner le jeu de données licornes dans l’ordre croissant des deux variables block et height :
p_care food block height weight mane_size fluffyness horn_rings
68 no_care high 1 1.2 18.24 16.6 148.1 7
86 no_care low 1 1.8 6.01 17.6 46.2 4
72 no_care high 1 2.1 19.15 15.6 176.7 6
84 no_care low 1 2.2 9.97 9.6 63.1 2
82 no_care low 1 2.3 7.28 13.8 32.8 6
56 no_care medium 1 2.5 14.85 17.5 77.8 10
69 no_care high 1 2.6 16.57 17.1 141.1 3
87 no_care low 1 3.0 9.93 12.0 56.6 6
53 no_care medium 1 3.5 12.93 16.6 109.3 3
88 no_care low 1 3.7 7.03 7.9 36.7 5Et si nous voulions ordonner licornes par ordre croissant selon la variable block mais par ordre décroissant de height? Nous pouvons utiliser une astuce simple en ajoutant un - avant l’argument licornes$height lorsque nous utilisons la fonction order(). Cela transformera essentiellement toutes les valeurs de height en négatives, ce qui aura pour effet d’inverser l’ordre. Notez que cette astuce ne fonctionne qu’avec des variables numériques.
bloc_invtaille_ord <- licornes[order(licornes$block, -licornes$height), ]
rbind(head(bloc_invtaille_ord, n = 10), tail(bloc_invtaille_ord, n = 10)) p_care food block height weight mane_size fluffyness horn_rings
21 care high 1 14.1 19.12 13.1 113.2 13
17 care high 1 12.6 18.66 18.6 54.0 9
38 care low 1 12.3 11.27 13.7 28.7 5
3 care medium 1 11.2 12.76 7.1 66.7 10
70 no_care high 1 10.9 17.22 49.2 189.6 17
2 care medium 1 10.7 12.14 14.1 46.0 10
4 care medium 1 10.4 8.78 11.9 20.3 1
5 care medium 1 10.4 13.58 14.5 26.9 4
22 care high 1 10.1 15.49 12.6 77.2 12
18 care high 1 10.0 18.07 16.9 90.5 3
64 no_care medium 2 3.9 12.97 17.0 97.5 5
93 no_care low 2 3.9 9.19 12.4 52.6 9
91 no_care low 2 3.7 8.10 10.5 60.5 6
94 no_care low 2 3.3 8.92 11.6 55.2 6
92 no_care low 2 3.2 7.45 14.1 38.1 4
42 care low 2 3.1 8.74 16.1 39.1 3
76 no_care high 2 2.6 18.88 16.4 181.5 14
89 no_care low 2 2.4 9.10 14.5 78.7 8
63 no_care medium 2 2.2 10.70 15.3 97.1 7
62 no_care medium 2 1.8 10.47 11.8 120.8 9Si nous voulons faire la même chose avec une variable de type facteur (ou caractère) comme food il faut utiliser la fonction xtfrm() sur cette variable, à l’intérieur de notre fonction order() :
invnourriture_taille_ord <- licornes[order(-xtfrm(licornes$food), licornes$height), ]
rbind(head(invnourriture_taille_ord, n = 10), tail(invnourriture_taille_ord, n = 10)) p_care food block height weight mane_size fluffyness horn_rings
62 no_care medium 2 1.8 10.47 11.8 120.8 9
63 no_care medium 2 2.2 10.70 15.3 97.1 7
56 no_care medium 1 2.5 14.85 17.5 77.8 10
53 no_care medium 1 3.5 12.93 16.6 109.3 3
58 no_care medium 2 3.9 9.07 9.6 90.4 7
64 no_care medium 2 3.9 12.97 17.0 97.5 5
52 no_care medium 1 4.1 12.58 13.9 136.6 11
16 care medium 2 4.5 12.55 13.4 14.4 6
60 no_care medium 2 4.5 13.68 14.8 125.5 9
55 no_care medium 1 4.9 6.89 8.2 52.9 3
29 care high 2 9.2 13.26 11.3 108.0 9
78 no_care high 2 9.3 18.75 18.4 181.1 16
18 care high 1 10.0 18.07 16.9 90.5 3
19 care high 1 10.0 13.29 15.8 142.7 12
22 care high 1 10.1 15.49 12.6 77.2 12
70 no_care high 1 10.9 17.22 49.2 189.6 17
25 care high 2 11.5 23.89 14.3 101.5 12
17 care high 1 12.6 18.66 18.6 54.0 9
21 care high 1 14.1 19.12 13.1 113.2 13
32 care high 2 17.2 19.20 10.9 89.9 14Il est à noter que la variable food a été classée dans l’ordre alphabétique inverse et que la variable height a été ordonnée par valeurs croissantes, à l’intérieur de chaque niveau de food.
Si nous voulions ordonner le jeu de données selon les niveau de la variable food, c.-à-d. low –> medium –> high au lieu de l’ordre alphabétique par défaut (high, low, medium), nous devons d’abord modifier l’ordre des niveaux de food dans le jeu de données à l’aide de la fonction factor(). Une fois que nous avons fait cela, nous pouvons utiliser la fonction order() comme d’habitude. Remarque : si vous lisez la version pdf de ce livre, la sortie a été tronquée pour économiser de l’espace.
licornes$food <- factor(licornes$food,
levels = c("low", "medium", "high")
)
nourriture_ord <- licornes[order(licornes$food), ]
rbind(head(nourriture_ord, n = 10), tail(nourriture_ord, n = 10)) p_care food block height weight mane_size fluffyness horn_rings
33 care low 1 8.0 6.88 9.3 16.1 4
34 care low 1 8.0 10.23 11.9 88.1 4
35 care low 1 6.4 5.97 8.7 7.3 2
36 care low 1 7.6 13.05 7.2 47.2 8
37 care low 1 9.7 6.49 8.1 18.0 3
38 care low 1 12.3 11.27 13.7 28.7 5
39 care low 1 9.1 8.96 9.7 23.8 3
40 care low 1 8.9 11.48 11.1 39.4 7
41 care low 2 7.4 10.89 13.3 9.5 5
42 care low 2 3.1 8.74 16.1 39.1 3
71 no_care high 1 7.2 15.21 15.9 135.0 14
72 no_care high 1 2.1 19.15 15.6 176.7 6
73 no_care high 2 4.7 13.42 19.8 124.7 5
74 no_care high 2 5.0 16.82 17.3 182.5 15
75 no_care high 2 6.5 14.00 10.1 126.5 7
76 no_care high 2 2.6 18.88 16.4 181.5 14
77 no_care high 2 6.0 13.68 16.2 133.7 2
78 no_care high 2 9.3 18.75 18.4 181.1 16
79 no_care high 2 4.6 14.65 16.7 91.7 11
80 no_care high 2 5.2 17.70 19.1 181.1 8Il est parfois utile de pouvoir ajouter des lignes et des colonnes de données supplémentaires à nos jeux de données. Il existe plusieurs façons d’y parvenir (comme toujours en R !) en fonction des circonstances. Pour ajouter simplement des lignes supplémentaires à un jeu de données existant, nous pouvons utiliser la fonction rbind() (row/‘ligne’ - bind/‘liaison’) et pour ajouter des colonnes, la fonction cbind() (columns/‘colonnes’ - bind/‘liaison’) . Créons quelques jeux de données test pour voir cela en action en utilisant notre vieille amie, la fonction data.frame() :
# rbind pour les lignes ('rows')
df1 <- data.frame(
id = 1:4, taille = c(120, 150, 132, 122),
poids = c(44, 56, 49, 45)
)
df1 id taille poids
1 1 120 44
2 2 150 56
3 3 132 49
4 4 122 45df2 <- data.frame(
id = 5:6, taille = c(119, 110),
poids = c(39, 35)
)
df2 id taille poids
1 5 119 39
2 6 110 35df3 <- data.frame(
id = 1:4, taille = c(120, 150, 132, 122),
poids = c(44, 56, 49, 45)
)
df3 id taille poids
1 1 120 44
2 2 150 56
3 3 132 49
4 4 122 45df4 <- data.frame(localisation = c("UK", "CZ", "CZ", "UK"))
df4 localisation
1 UK
2 CZ
3 CZ
4 UKNous pouvons utiliser la fonction rbind() pour ajouter les lignes du jeu de données df2 aux lignes de df1 et créer le nouveau jeu de données à df_lcomb :
df_lcomb <- rbind(df1, df2)
df_lcomb id taille poids
1 1 120 44
2 2 150 56
3 3 132 49
4 4 122 45
5 5 119 39
6 6 110 35Et cbind pour ajouter la colonne de df4 à la colonne df3 et l’assigner à df_ccomb :
df_ccomb <- cbind(df3, df4)
df_ccomb id height weight location
1 1 120 44 UK
2 2 150 56 CZ
3 3 132 49 CZ
4 4 122 45 UKUne autre situation dans laquelle l’ajout d’une nouvelle colonne à un jeu de données est utile est lorsque vous souhaitez effectuer une sorte de transformation sur une variable existante. Par exemple, supposons que nous voulions appliquer une transformation logarithmique, log10 à une variable existante, la variable taille dans le jeu de données df_rcomb que nous avons créée ci-dessus. Nous pourrions simplement créer une variable distincte contenant ces valeurs, mais il est préférable de créer cette variable directement en tant que nouvelle colonne dans notre jeu de données existant afin de conserver toutes nos données ensemble. Appelons cette nouvelle variable taille_log10.
# transformation log10
df_lcomb$taille_log10 <- log10(df_lcomb$taille)
df_lcomb id taille poids taille_log10
1 1 120 44 2.079181
2 2 150 56 2.176091
3 3 132 49 2.120574
4 4 122 45 2.086360
5 5 119 39 2.075547
6 6 110 35 2.041393Cette situation survient également lorsque nous voulons changer le type d’une variable existante dans un jeu de données. Par exemple, la variable id dans la base de données df_lcomb est une donnée de type numérique (utilisez la fonction str() ou class() pour le vérifier). Si nous voulions convertir id en un facteur à utiliser plus tard dans notre analyse, nous pouvons créer une nouvelle variable appelée id_f dans notre jeu de données et utiliser la fonction factor() pour convertir la variable id.
# conversion en un facteur
df_lcomb$id_f <- factor(df_lcomb$id)
df_lcomb id taille poids taille_log10 id_f
1 1 120 44 2.079181 1
2 2 150 56 2.176091 2
3 3 132 49 2.120574 3
4 4 122 45 2.086360 4
5 5 119 39 2.075547 5
6 6 110 35 2.041393 6str(df_lcomb)'data.frame': 6 obs. of 5 variables:
$ id : int 1 2 3 4 5 6
$ taille : num 120 150 132 122 119 110
$ poids : num 44 56 49 45 39 35
$ taille_log10: num 2.08 2.18 2.12 2.09 2.08 ...
$ id_f : Factor w/ 6 levels "1","2","3","4",..: 1 2 3 4 5 6Au lieu d’ajouter simplement des lignes ou des colonnes à un jeu de données, nous pouvons également fusionner deux jeux de données. Supposons que nous ayons un jeu de données contenant des informations taxonomiques sur certains invertébrés communs des côtes rocheuses du Royaume-Uni (appelé taxa) et un autre jeu de données qui contient des informations sur l’endroit où ils se trouvent habituellement sur le littoral rocheux (appelé zone). Nous pouvons fusionner ces jeux cadres de données pour produire un seul jeu de données contenant à la fois des informations taxonomiques et des informations sur l’emplacement. Commençons par créer ces deux jeux de données (en réalité, il vous suffira probablement d’importer vos différents ensembles de données).
taxa <- data.frame(
GENUS = c("Patella", "Littorina", "Halichondria", "Semibalanus"),
espece = c("vulgata", "littoria", "panacea", "balanoides"),
famille = c("patellidae", "Littorinidae", "Halichondriidae", "Archaeobalanidae")
)
taxa GENUS espece famille
1 Patella vulgata patellidae
2 Littorina littoria Littorinidae
3 Halichondria panacea Halichondriidae
4 Semibalanus balanoides Archaeobalanidaezone <- data.frame(
genus = c(
"Laminaria", "Halichondria", "Xanthoria", "Littorina",
"Semibalanus", "Fucus"
),
espece = c(
"digitata", "panacea", "parietina", "littoria",
"balanoides", "serratus"
),
zone = c("v_bas", "bas", "v_haut", "bas_moy", "haut", "bas_moy")
)
zone genus espece zone
1 Laminaria digitata v_bas
2 Halichondria panacea bas
3 Xanthoria parietina v_haut
4 Littorina littoria bas_moy
5 Semibalanus balanoides haut
6 Fucus serratus bas_moyPuisque nos deux jeux de données contiennent au moins une variable en commun (espece dans notre cas), nous pouvons simplement utiliser la fonction merge() (‘fusionner’) pour créer un nouveau jeu de données appelé taxa_zone.
taxa_zone <- merge(x = taxa, y = zone)
taxa_zone species GENUS family genus zone
1 balanoides Semibalanus Archaeobalanidae Semibalanus high
2 littoria Littorina Littorinidae Littorina low_mid
3 panacea Halichondria Halichondriidae Halichondria lowRemarquez que le jeu de données fusionné ne contient que les lignes ayant des espèces dans les 2 jeux de données. Il existe également deux colonnes appelées GENUS et genus car la fonction merge() les traite comme deux variables différentes provenant des deux jeux de données.
Si nous voulons inclure toutes les données des deux jeux de données, nous devrons utiliser l’argument all = TRUE dans la fonction merge(). Les valeurs manquantes seront incluses en tant que NA :
taxa_zone <- merge(x = taxa, y = zone, all = TRUE)
taxa_zone species GENUS family genus zone
1 balanoides Semibalanus Archaeobalanidae Semibalanus high
2 digitata <NA> <NA> Laminaria v_low
3 littoria Littorina Littorinidae Littorina low_mid
4 panacea Halichondria Halichondriidae Halichondria low
5 parietina <NA> <NA> Xanthoria v_high
6 serratus <NA> <NA> Fucus low_mid
7 vulgata Patella patellidae <NA> <NA>Si les noms des variables sur lesquelles vous souhaitez baser la fusion sont différents dans chaque jeux de données (par exemple GENUS et genus), vous pouvez spécifier les noms dans le premier jeu de données (appelé x) et dans le second jeu de données (appelé y) à l’aide des arguments by.x = et by.y =.
taxa_zone <- merge(x = taxa, y = zone, by.x = "GENUS", by.y = "genus", all = TRUE)
taxa_zone GENUS species.x family species.y zone
1 Fucus <NA> <NA> serratus low_mid
2 Halichondria panacea Halichondriidae panacea low
3 Laminaria <NA> <NA> digitata v_low
4 Littorina littoria Littorinidae littoria low_mid
5 Patella vulgata patellidae <NA> <NA>
6 Semibalanus balanoides Archaeobalanidae balanoides high
7 Xanthoria <NA> <NA> parietina v_highOu utiliser plusieurs noms de variables :
taxa_zone <- merge(
x = taxa, y = zone, by.x = c("espece", "GENUS"),
by.y = c("espece", "genus"), all = TRUE
)
taxa_zone espece GENUS famille zone
1 balanoides Semibalanus Archaeobalanidae haut
2 digitata Laminaria <NA> v_bas
3 littoria Littorina Littorinidae bas_moy
4 panacea Halichondria Halichondriidae bas
5 parietina Xanthoria <NA> v_haut
6 serratus Fucus <NA> bas_moy
7 vulgata Patella patellidae <NA>Le remodelage des données dans différents formats est une tâche courante. Avec des données de type rectangulaire (les jeux de données ont le même nombre de lignes dans chaque colonne), vous rencontrerez deux formes principales de jeu de données : le format “long” (parfois appelé “empilé”) et le format “large”. Un exemple de jeu de données au format “long” est donné ci-dessous. Nous pouvons voir que chaque ligne représente une observation unique d’un sujet individuel et que chaque sujet peut avoir plusieurs lignes. Il en résulte une seule colonne de notre mesures.
donnees_longue <- data.frame(
sujet = rep(c("A", "B", "C", "D"), each = 3),
sexe = rep(c("M", "F", "F", "M"), each = 3),
condition = rep(c("controle", "cond1", "cond2"), times = 4),
mesures = c(
12.9, 14.2, 8.7, 5.2, 12.6, 10.1, 8.9,
12.1, 14.2, 10.5, 12.9, 11.9
)
)
donnees_longue sujet sexe condition mesures
1 A M controle 12.9
2 A M cond1 14.2
3 A M cond2 8.7
4 B F controle 5.2
5 B F cond1 12.6
6 B F cond2 10.1
7 C F controle 8.9
8 C F cond1 12.1
9 C F cond2 14.2
10 D M controle 10.5
11 D M cond1 12.9
12 D M cond2 11.9Nous pouvons également formater les mêmes données au format “large”, comme indiqué ci-dessous. Dans ce format, nous avons plusieurs observations de chaque sujet dans une seule ligne avec des mesures dans différentes colonnes (controle, cond1 et cond2). Il s’agit d’un format courant lorsqu’il s’agit de mesures répétées à partir d’unités d’échantillonnage.
donnees_large <- data.frame(
sujet = c("A", "B", "C", "D"),
sexe = c("M", "F", "F", "M"),
controle = c(12.9, 5.2, 8.9, 10.5),
cond1 = c(14.2, 12.6, 12.1, 12.9),
cond2 = c(8.7, 10.1, 14.2, 11.9)
)
donnees_large sujet sexe controle cond1 cond2
1 A M 12.9 14.2 8.7
2 B F 5.2 12.6 10.1
3 C F 8.9 12.1 14.2
4 D M 10.5 12.9 11.9Bien qu’il n’y ait pas de problème inhérent à l’un ou l’autre de ces formats, il est parfois nécessaire d’effectuer une conversion entre les deux, car certaines fonctions requièrent un format spécifique pour fonctionner. Le format le plus courant est le format “long”.
Il existe de nombreuses façons de convertir ces deux formats, mais nous utiliserons les fonctions melt() et dcast() du paquet reshape2 📦 (vous devez d’abord l’installer). Les fonctions melt() est utilisée pour convertir les formats larges en formats longs. Le premier argument de la fonction melt() est la base de données que nous voulons faire fondre (dans notre cas wide_data). La fonction id.vars = c("subject", "sex") est un vecteur des variables que vous souhaitez empiler, l’argument measured.vars = c("control", "cond1", "cond2") identifie les colonnes des mesures dans les différentes conditions, l’argument variable.name = "condition" spécifie ce que vous voulez appeler la colonne empilée de vos différentes conditions dans votre cadre de données de sortie et l’argument value.name = "measurement" est le nom de la colonne de vos mesures empilées dans votre cadre de données de sortie.
sujet sexe controle cond1 cond2
1 A M 12.9 14.2 8.7
2 B F 5.2 12.6 10.1
3 C F 8.9 12.1 14.2
4 D M 10.5 12.9 11.9# conversion du "large" en "long"
mon_df_long <- melt(
data = donnees_large, id.vars = c("sujet", "sexe"),
vars.mesures = c("controle", "cond1", "cond2"),
nom.variable = "condition", value.name = "mesures"
)
mon_df_long sujet sexe variable mesures
1 A M controle 12.9
2 B F controle 5.2
3 C F controle 8.9
4 D M controle 10.5
5 A M cond1 14.2
6 B F cond1 12.6
7 C F cond1 12.1
8 D M cond1 12.9
9 A M cond2 8.7
10 B F cond2 10.1
11 C F cond2 14.2
12 D M cond2 11.9La fonction dcast() est utilisée pour convertir un jeu de données au format “long” en un jeu de données au format “large”. Le premier argument est à nouveau le jeu de données que nous voulons convertir (donnees_longue pour cet exemple). Le deuxième argument est la syntaxe de la formule. L’argument sujet + sexe de la formule signifie que nous voulons garder ces colonnes séparées, et l’élément ~ condition est la colonne qui contient les étiquettes que nous voulons diviser en nouvelles colonnes dans notre nouveau jeu de données. La colonne var.valeur = "mesures" est la colonne qui contient les données mesurées.
donnees_longue # rappelons-nous à quoi ressemble le format "long" sujet sexe condition mesures
1 A M controle 12.9
2 A M cond1 14.2
3 A M cond2 8.7
4 B F controle 5.2
5 B F cond1 12.6
6 B F cond2 10.1
7 C F controle 8.9
8 C F cond1 12.1
9 C F cond2 14.2
10 D M controle 10.5
11 D M cond1 12.9
12 D M cond2 11.9# conversion du "long" en "large"
mon_df_large <- dcast(
data = donnees_longue, sujet + sexe ~ condition,
var.valeur = "mesures"
)
mon_df_large sujet sexe cond1 cond2 controle
1 A M 14.2 8.7 12.9
2 B F 12.6 10.1 5.2
3 C F 12.1 14.2 8.9
4 D M 12.9 11.9 10.5tidyverse (L’univers ordonné)il semble que ce ne soit pas très ordonné ici et que nous devons améliorer ça.
Maintenant que nous sommes en mesure de manipuler et extraire des données de nos jeux de données, notre prochaine tâche est de commencer à explorer et connaître nos données. Dans cette section, nous commencerons à produire des tableaux de statistiques récapitulatives utiles sur les variables de notre jeu de données et, dans les deux chapitres suivants, nous aborderons la visualisation de nos données à l’aide de graphiques R de base et l’utilisation du paquet ggplot2 📦.
Un point de départ très utile consiste à produire des statistiques récapitulatives simples pour toutes les variables de notre jeu de données licornes à l’aide de la fonction summary() :
summary(licornes) p_care food block height weight
Length:96 low :32 Min. :1.0 Min. : 1.200 Min. : 5.790
Class :character medium:32 1st Qu.:1.0 1st Qu.: 4.475 1st Qu.: 9.027
Mode :character high :32 Median :1.5 Median : 6.450 Median :11.395
Mean :1.5 Mean : 6.840 Mean :12.155
3rd Qu.:2.0 3rd Qu.: 9.025 3rd Qu.:14.537
Max. :2.0 Max. :17.200 Max. :23.890
mane_size fluffyness horn_rings
Min. : 5.80 Min. : 5.80 Min. : 1.000
1st Qu.:11.07 1st Qu.: 39.05 1st Qu.: 4.000
Median :13.45 Median : 70.05 Median : 6.000
Mean :14.05 Mean : 79.78 Mean : 7.062
3rd Qu.:16.45 3rd Qu.:113.28 3rd Qu.: 9.000
Max. :49.20 Max. :189.60 Max. :17.000 Pour les variables numériques (c’est-à-dire height, weight etc.), la moyenne, le minimum, le maximum, la médiane, le premier quartile (inférieur) et le troisième quartile (supérieur) sont présentés. Pour les variables factorielles (i.e. care et food), le nombre d’observations dans chacun des niveaux est indiqué. Si une variable contient des données manquantes, le nombre de NA est également indiqué.
Si nous voulons résumer un sous-ensemble plus petit de variables dans notre jeu de données, nous pouvons utiliser nos compétences en matière d’indexation en combinaison avec la fonction summary(). Par exemple, pour résumer uniquement les variables height, weight, mane_size et fluffyness nous pouvons inclure les index de colonne appropriés avec les crochets [ ]. Remarquez que nous incluons toutes les lignes en ne spécifiant pas d’index de ligne :
summary(licornes[, 4:7]) height weight mane_size fluffyness
Min. : 1.200 Min. : 5.790 Min. : 5.80 Min. : 5.80
1st Qu.: 4.475 1st Qu.: 9.027 1st Qu.:11.07 1st Qu.: 39.05
Median : 6.450 Median :11.395 Median :13.45 Median : 70.05
Mean : 6.840 Mean :12.155 Mean :14.05 Mean : 79.78
3rd Qu.: 9.025 3rd Qu.:14.537 3rd Qu.:16.45 3rd Qu.:113.28
Max. :17.200 Max. :23.890 Max. :49.20 Max. :189.60 # ou de manière équivalente
# summary(licornes[, c("height", "weight", "mane_size", "fluffyness")])Et pour résumer une seule variable :
summary(licornes$mane_size) Min. 1st Qu. Median Mean 3rd Qu. Max.
5.80 11.07 13.45 14.05 16.45 49.20 # ou de manière équivalente
# summary(licornes[, 6])Comme vous l’avez vu plus haut, le summary() indique le nombre d’observations dans chaque niveau de nos variables factorielles. Une autre fonction utile pour générer des tableaux de comptage est la fonction table(). La fonction table() peut être utilisée pour construire des tables de contingence de différentes combinaisons de niveaux de facteurs. Par exemple, pour compter le nombre d’observations pour chaque niveau de food :
table(licornes$food)
low medium high
32 32 32 Nous pouvons aller plus loin en produisant un tableau des effectifs pour chaque combinaison des niveau de food et care :
table(licornes$food, licornes$p_care)
care no_care
low 16 16
medium 16 16
high 16 16Une version plus souple de la fonction table() est la fonction xtabs(). La fonction xtabs() utilise une notation de formule (~) pour construire des tables de contingence avec les variables de classification croisée séparées par un + à droite de la formule. xtabs() contient également un argument utile, data =, pour ne pas avoir à inclure le nom du jeu de données lors de la spécification de chaque variable :
xtabs(~ food + p_care, data = licornes) p_care
food care no_care
low 16 16
medium 16 16
high 16 16Nous pouvons même construire des tables de contingence plus compliquées en utilisant davantage de variables. Notez que dans l’exemple ci-dessous, la variable xtabs() a discrètement contraint notre block à être un facteur.
xtabs(~ food + p_care + block, data = licornes), , block = 1
p_care
food care no_care
low 8 8
medium 8 8
high 8 8
, , block = 2
p_care
food care no_care
low 8 8
medium 8 8
high 8 8Et pour un tableau mieux formaté, nous pouvons imbriquer la fonction xtabs() à l’intérieur de la fonction ftable() pour “aplatir” le tableau.
block 1 2
food p_care
low care 8 8
no_care 8 8
medium care 8 8
no_care 8 8
high care 8 8
no_care 8 8Nous pouvons également résumer nos données pour chaque niveau d’une variable factorielle. Supposons que nous voulions calculer la valeur moyenne de height pour chacun de nos niveaux low, meadium et high de la variable food. Pour ce faire, nous utilisons la fonction mean() que nous appliquons à la variable height pour chaque niveau de food en utilisant la fonction tapply().
tapply(licornes$height, licornes$food, mean) low medium high
5.853125 7.012500 7.653125 La fonction tapply() n’est pas limitée au calcul des valeurs moyennes, vous pouvez l’utiliser pour appliquer de nombreuses fonctions fournies avec R ou même des fonctions que vous avez écrites vous-même (voir le Chapitre 5 pour plus de détails). Par exemple, nous pouvons appliquer la fonction sd() pour calculer l’écart-type pour chaque niveau de food ou même la fonction summary().
tapply(licornes$height, licornes$food, sd) low medium high
2.828425 3.005345 3.483323 tapply(licornes$height, licornes$food, summary)$low
Min. 1st Qu. Median Mean 3rd Qu. Max.
1.800 3.600 5.550 5.853 8.000 12.300
$medium
Min. 1st Qu. Median Mean 3rd Qu. Max.
1.800 4.500 7.000 7.013 9.950 12.300
$high
Min. 1st Qu. Median Mean 3rd Qu. Max.
1.200 5.800 7.450 7.653 9.475 17.200 Remarque : si la variable que vous souhaitez résumer contient des valeurs manquantes (NA), vous devrez également inclure un argument spécifiant comment vous souhaitez que la fonction traite les valeurs manquantes (NA). Nous en avons vu un exemple dans la Section 2.5.5 où la fonction mean() a renvoyé une valeur NA en cas de données manquantes. Pour inclure les NA, il suffit d’ajouter l’argument na.rm = TRUE lors de l’utilisation de tapply().
tapply(licornes$height, licornes$food, mean, na.rm = TRUE) low medium high
5.853125 7.012500 7.653125 Nous pouvons également utiliser tapply() pour appliquer des fonctions à plus d’un seul facteur. La seule chose à retenir est que les facteurs doivent être fournis à la fonction tapply() sous la forme d’une liste à l’aide de la fonction list(). Pour calculer la moyenne de height pour chaque combinaison de food et care nous pouvons utiliser la notation list(licornes$food, licornes$p_care).
care no_care
low 8.0375 3.66875
medium 9.1875 4.83750
high 9.6000 5.70625Et si vous en avez un peu marre d’avoir à écrire licornes$ pour chaque variable, vous pouvez imbriquer la notation tapply() à l’intérieur de la fonction with(). La fonction with() permet à R d’évaluer une expression par rapport à un objet de données nommé (dans ce cas, le jeu de données licornes).
care no_care
low 8.0375 3.66875
medium 9.1875 4.83750
high 9.6000 5.70625La fonction with() fonctionne également avec de nombreuses autres fonctions et peut vous faire économiser beaucoup de temps de frappe !
Une autre fonction très utile pour résumer des données est la fonction aggregate(). La fonction aggregate() fonctionne de manière très similaire à la fonction tapply() mais est un peu plus flexible.
Par exemple, pour calculer la moyenne des variables height, weight, mane_size et fluffyness pour chaque niveau de food :
food height weight mane_size fluffyness
1 low 5.853125 8.652812 11.14375 45.1000
2 medium 7.012500 11.164062 13.83125 67.5625
3 high 7.653125 16.646875 17.18125 126.6875Dans le code ci-dessus, nous avons indexé les colonnes que nous voulons résumer dans le jeu de données licornes à l’aide de la notation licornes[, 4:7]. Les by = (“par =”) spécifie une liste de facteurs (list(food = licornes$food)) et l’argument FUN = désigne la fonction à appliquer (mean dans cet exemple).
Comme dans le cas de la fonction tapply() nous pouvons inclure plus d’un facteur sur lequel appliquer une fonction. Ici, nous calculons les valeurs moyennes pour chaque combinaison de food et care :
food p_care height weight mane_size fluffyness
1 low care 8.03750 9.016250 9.96250 30.30625
2 medium care 9.18750 11.011250 13.48750 40.59375
3 high care 9.60000 16.689375 15.54375 98.05625
4 low no_care 3.66875 8.289375 12.32500 59.89375
5 medium no_care 4.83750 11.316875 14.17500 94.53125
6 high no_care 5.70625 16.604375 18.81875 155.31875Nous pouvons également utiliser la fonction aggregate() d’une manière différente en utilisant la méthode de la formule (comme nous l’avons fait avec la fonction xtabs()). Dans la partie gauche de la formule (~), nous spécifions la variable à laquelle nous voulons appliquer la fonction mean() et, dans la partie droite, nos facteurs séparés par un symbole +. La méthode de la formule permet également d’utiliser l’argument data = pour des raisons de commodité.
aggregate(height ~ food + p_care, FUN = mean, data = licornes) food p_care height
1 low care 8.03750
2 medium care 9.18750
3 high care 9.60000
4 low no_care 3.66875
5 medium no_care 4.83750
6 high no_care 5.70625L’un des avantages de la méthode de la formule est qu’elle permet également d’utiliser l’argument subset = pour appliquer la fonction à des sous-ensembles des données originales. Par exemple, pour calculer la moyenne de height pour chaque combinaison de food et care mais seulement pour les licornes qui ont moins de 7 anneaux sur leur corne (horn_rings).
aggregate(height ~ food + p_care, FUN = mean, subset = horn_rings < 7, data = licornes) food p_care height
1 low care 8.176923
2 medium care 8.570000
3 high care 7.900000
4 low no_care 3.533333
5 medium no_care 5.316667
6 high no_care 3.850000Ou seulement pour les licornes du block 1.
aggregate(height ~ food + p_care, FUN = mean, subset = block == "1", data = licornes) food p_care height
1 low care 8.7500
2 medium care 9.5375
3 high care 10.0375
4 low no_care 3.3250
5 medium no_care 5.2375
6 high no_care 5.9250Nous espérons que vous avez maintenant une idée de la puissance et de l’utilité de R pour manipuler et résumer des données (et nous n’avons fait qu’effleurer la surface). L’un des grands avantages de l’utilisation de R pour manipuler vos données est que vous disposez d’un enregistrement permanent de tout ce que vous avez fait avec vos données. Fini le temps des modifications non documentées dans Excel ou Calc ! En traitant vos données en “lecture seule” et en documentant toutes vos décisions dans R, vous aurez fait un grand pas en avant pour rendre votre analyse plus reproductible et plus transparente pour les autres. Il est toutefois important de savoir que les modifications apportées à votre base de données dans R ne changeront pas le fichier de données original que vous avez importé dans R (et c’est une bonne chose). Heureusement, il est facile d’exporter des cadres de données vers des fichiers externes dans une grande variété de formats.
La principale fonction d’exportation de jeux de données est write.table(). Comme pour la fonction read.table() la fonction write.table() est très flexible et dispose de nombreux arguments permettant de personnaliser son comportement. Prenons l’exemple de notre jeu de données d’origine licornes et exportons ces modifications vers un fichier externe.
De la même manière que pour read.table(), write.table() possède une série de fonctions avec des valeurs par défaut spécifiques au format, telles que write.csv() et write.delim() qui utilisent respectivement “,” et les tabulations comme délimiteurs, et incluent les noms de colonnes par défaut.
Ordonnons les lignes du jeu de données par ordre croissant de la variable height à l’intérieur de chaque niveau de food. Nous appliquerons également une transformation racine carrée à la variable du nombre d’anneaux de la corne (horn_rings) et une transformation logarithmique, log10 sur la variable height et enregistrons ça en tant que colonnes supplémentaires dans notre jeu de données (nous espérons que cela vous est familier maintenant !).
licornes_df2 <- licornes[order(licornes$food, licornes$height), ]
licornes_df2$horn_rings_sqrt <- sqrt(licornes_df2$horn_rings)
licornes_df2$log10_height <- log10(licornes_df2$height)
str(licornes_df2)'data.frame': 96 obs. of 10 variables:
$ p_care : chr "no_care" "no_care" "no_care" "no_care" ...
$ food : Factor w/ 3 levels "low","medium",..: 1 1 1 1 1 1 1 1 1 1 ...
$ block : int 1 1 1 2 1 2 2 2 1 2 ...
$ height : num 1.8 2.2 2.3 2.4 3 3.1 3.2 3.3 3.7 3.7 ...
$ weight : num 6.01 9.97 7.28 9.1 9.93 8.74 7.45 8.92 7.03 8.1 ...
$ mane_size : num 17.6 9.6 13.8 14.5 12 16.1 14.1 11.6 7.9 10.5 ...
$ fluffyness : num 46.2 63.1 32.8 78.7 56.6 39.1 38.1 55.2 36.7 60.5 ...
$ horn_rings : int 4 2 6 8 6 3 4 6 5 6 ...
$ horn_rings_sqrt: num 2 1.41 2.45 2.83 2.45 ...
$ log10_height : num 0.255 0.342 0.362 0.38 0.477 ...Nous pouvons maintenant exporter notre nouveau jeu de données licornes_df2 à l’aide de la fonction write.table(). Le premier argument est le jeu de données que vous voulez exporter (licornes_df2 dans notre exemple). Nous donnons ensuite le nom du fichier que nous voulons créer (avec son extension) et le chemin d’accès au fichier entre guillemets simples ou doubles en utilisant l’argument file =. Dans cet exemple, nous exportons le jeu de données vers un fichier appelé licornes_transformee.csv dans le répertoire donnees. L’argument row.names = FALSE empêche R d’inclure les noms des lignes dans la première colonne du fichier.
write.csv(licornes_transformee,
file = "donnees/licornes_transformee.csv",
row.names = FALSE
)Comme nous avons enregistré le fichier en tant que fichier texte délimité par des virgules (CSV), nous pouvons ouvrir ce fichier dans n’importe quel éditeur de texte.
Nous pouvons bien sûr exporter nos fichiers dans une variété d’autres formats.
Comme pour l’importation de fichiers de données dans R, il existe également de nombreuses fonctions alternatives pour exporter des données vers des fichiers externes, en plus de la fonction write.table(). Si vous avez suivi la rubrique “Autres fonctions d’importation” Section 3.3.4 de ce chapitre, vous aurez déjà installé les paquets nécessaires.
La fonction fwrite() du paquet data.table 📦 est très efficace pour exporter des objets de données volumineux et est beaucoup plus rapide que la fonction write.table(). Il est également assez simple à utiliser car il possède la plupart des arguments de la fonction write.table(). Pour exporter un fichier texte délimité par des tabulations, il suffit de spécifier le nom du jeu de données, le nom et le chemin du fichier de sortie et le séparateur entre les colonnes :
library(data.table)
fwrite(licornes_df2, file = "donnees/licornes_04_12.txt", sep = "\t")Pour exporter un fichier csv délimité, c’est encore plus facile car nous n’avons même pas besoin d’inclure l’option sep = :
library(data.table)
fwrite(licornes_df2, file = "donnees/licornes_04_12.csv")Le paquet readr 📦 est également fourni avec deux fonctions utiles pour exporter rapidement des données dans des fichiers externes : la fonction write_tsv() pour écrire des fichiers délimités par des tabulations et la fonction write_csv() pour enregistrer des fichiers de valeurs séparées par des virgules (csv).
Pour des raisons de place et de simplicité, nous ne montrons que les cinq premières et cinq dernières lignes.↩︎