1 + 1[1] 2Objectif de ce chapitre :
Les captures d’écrans présentées proviennent de RStudio mais tout est très similaire sur VSCode.
Avant de poursuivre, quelques points à garder à l’esprit tout au long de ce chapitre :
R est sensible à la casse, c’est-à-dire que A n’est pas la même chose que a et anova, ce n’est pas Anova.
Tout ce qui suit un # est interprété comme un commentaire et ignoré par R. Ces commentaires doivent être utilisés librement dans votre code, à la fois pour votre propre information et pour aider vos collaborateurs. L’écriture de commentaires est un peu un art que vous maîtriserez de mieux en mieux avec l’expérience.
Dans R, les commandes sont généralement séparées par une nouvelle ligne. Vous pouvez également utiliser un point-virgule ; pour séparer vos commandes, mais nous vous le déconseillons fortement (rend le code très difficilement lisible).
Si une invite de continuation + apparaît dans la console après l’exécution de votre code, cela signifie que vous n’avez pas terminé votre code correctement. Cela se produit souvent lorsque vous oubliez de fermer une parenthèse, ce qui est particulièrement fréquent lors que l’on utilise des parenthèses imbriquées ((((commande quelconque))). Terminez simplement la commande sur la nouvelle ligne ou appuyez sur la touche “escape” de votre clavier (voir le point ci-dessous) et corrigez la faute de frappe.
En général, R est assez tolérant vis-à-vis des espaces supplémentaires insérés dans votre code, en fait l’utilisation d’espaces est activement encouragée. Cependant, les espaces ne doivent pas être insérés dans les opérateurs, c’est-à-dire <- ne peut pas s’écrire < - (notez l’espace). Voir le guide de style pour savoir où placer les espaces afin de rendre votre code plus lisible.
Si votre console se bloque et ne répond plus après l’exécution d’une commande, vous pouvez souvent vous sortir d’affaire en appuyant sur la touche “escape” (esc) de votre clavier ou en cliquant sur l’icône d’arrêt/stop en haut à droite de votre console. Cela mettra fin à la plupart des opérations en cours.
Dans le Chapitre 1, nous avons appris ce qu’était la console R la création de scripts et de Projets. Nous avons également vu comment écrire votre code R dans un script, puis comment insérer ce code dans la console pour qu’il s’exécute (si vous avez oublié comment faire, revenez à la section sur la console (1.2.1.1) pour vous rafraîchir la mémoire). Le fait d’écrire votre code dans un script signifie que vous aurez un enregistrement permanent de tout ce que vous avez fait (à condition de sauvegarder votre script) et vous permet également de faire de nombreux commentaires pour vous rappeler ce que vous aviez fait (ou voulu faire) quand vous retournerez si votre code à l’avenir. Ainsi, pendant que vous travaillez sur ce chapitre, nous vous suggérons de créer un nouveau script (ou un Projet Rstudio) pour écrire votre code au fur et à mesure.
Comme nous l’avons vu au Chapitre 1, nous pouvons utiliser R de la même manière qu’une calculatrice. Nous pouvons saisir une expression arithmétique dans notre script, puis l’envoyer dans la console et recevoir un résultat. Par exemple, si nous tapons l’expression 1 + 1 et que l’on exécute cette ligne de code dans la console, on obtient la réponse 2 (😃 !)
1 + 1[1] 2Le [1] devant le résultat indique que l’observation au début de la ligne est la première. Cela n’est pas très utile dans cet exemple, mais peut l’être lors de l’impression de résultats sur plusieurs lignes (nous en verrons un exemple ci-dessous). Les autres opérateurs arithmétiques évidents sont -, *, / pour la soustraction, la multiplication et la division respectivement. Pour la multiplication de la matrice, l’opérateur est %*%.
R suit la convention mathématique habituelle de l’ordre des opérations. Par exemple, l’expression 2 + 3 * 4 est interprétée comme ayant la valeur 2 + (3 * 4) = 14 et non (2 + 3) * 4 = 20. Il existe un grand nombre de fonctions mathématiques dans R, dont les plus utiles sont les suivantes : log(), log10(), exp(), sqrt().
log(1) # logarithme en base e[1] 0log10(1) # logarithme en base 10[1] 0exp(1) # antilog naturel, fonction exponentielle[1] 2.718282sqrt(4) # racine carrée[1] 24^2 # 4 puissance 2[1] 16pi # pas une fonction mais utile[1] 3.141593Il est important de comprendre que lorsque vous exécutez un code comme nous l’avons fait ci-dessus, le résultat du code (ou valeur) n’est affiché que dans la console. Bien que cela puisse parfois être utile, il est généralement beaucoup plus pratique de stocker la ou les valeurs dans un objet.
Au cœur de presque tout ce que vous ferez (ou ferez probablement) en R se trouve le concept selon lequel tout en R est un objet. Ces objets peuvent être pratiquement n’importe quoi, d’un simple nombre ou d’une chaîne de caractères (comme un mot) à des structures très complexes comme la sortie d’un graphique, un résumé de votre analyse statistique ou un ensemble de commandes R effectuant une tâche spécifique. Pour comprendre R, il est essentiel de savoir comment créer des objets et leur attribuer des valeurs.
Pour créer un objet, il suffit de lui donner un nom. Nous pouvons ensuite attribuer une valeur à cet objet à l’aide d’un opérateur d’affectation <- (parfois appelé opérateur d’obtention). L’opérateur d’affectation est un symbole composite composé d’un symbole “moins que” < et d’un trait d’union -
raccourci clavier : “option” + “-” sur Mac ; “alt” + “-” sur Windows.
mon_obj <- 32Dans le code ci-dessus, nous avons créé un objet appelé mon_obj et lui avons attribué la valeur numérique 32 à l’aide de l’opérateur d’affectation (dans notre tête, nous lisons toujours cela comme ‘mon_obj est 32’). Vous pouvez également utiliser = à la place de <- pour assigner des valeurs, mais c’est une mauvaise pratique car cela peut entraîner des confusions plus tard quand vous programmerez en R (voir Chapitre 5) donc nous vous déconseillons d’utiliser cette notation.
Pour afficher la valeur de l’objet, il suffit de taper son nom.
mon_obj[1] 32Maintenant que nous avons créé cet objet, R le connaît et en gardera la trace pendant la session R en cours. Tous les objets que vous créez sont stockés dans l’espace de travail actuel et vous pouvez visualiser tous les objets de votre espace de travail dans RStudio en cliquant sur l’onglet “Environnement” dans le volet supérieur droit.
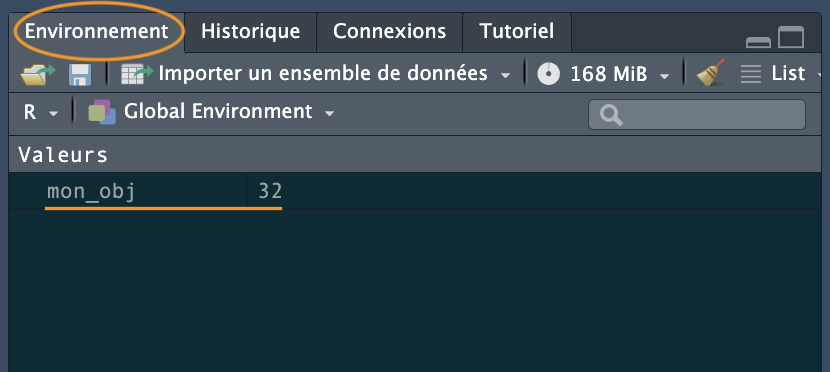
Si vous cliquez sur la flèche vers le bas de l’icône “List” (Liste) dans le même volet et que vous passez à l’affichage “Grid” (Grille), RStudio vous présentera un résumé des objets, y compris le type (“numeric” (numérique) - c’est un nombre), la longueur (une seule valeur dans cet objet), sa taille “physique” et sa valeur (32 dans ce cas). Dans VSCode, allez sur le panneau d’extension R et vous obtiendrez les mêmes informations.
Il existe de nombreux types de valeurs que vous pouvez attribuer à un objet. Par exemple
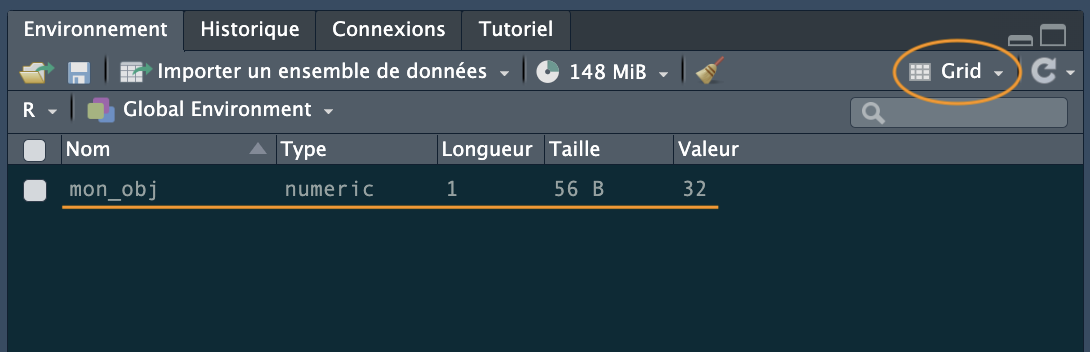
mon_obj2 <- "R c'est trop bien"Nous avons créé un objet appelé mon_obj2 et lui avons attribué la valeur R c'est trop bien qui est une chaîne de caractères. Remarquez que nous avons mis la chaîne de caractères entre guillemets. Si vous oubliez d’utiliser les guillemets, vous recevrez un message d’erreur.
Notre espace de travail contient maintenant les deux objets que nous avons créés jusqu’à présent avec mon_obj2 de type “character” (caractère).
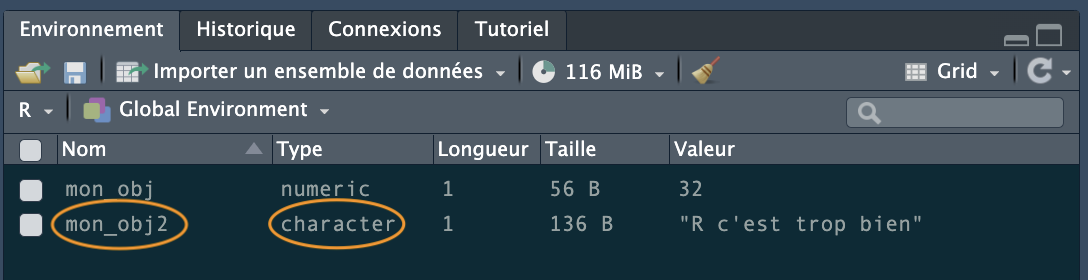
Pour modifier la valeur d’un objet existant, il suffit de lui réattribuer une nouvelle valeur. Par exemple, pour modifier la valeur de mon_obj2 de "R c'est trop bien" au nombre 1024
mon_obj2 <- 1024Remarquez que le type est devenu numérique et que la valeur est passée à 1024 dans l’environnement.
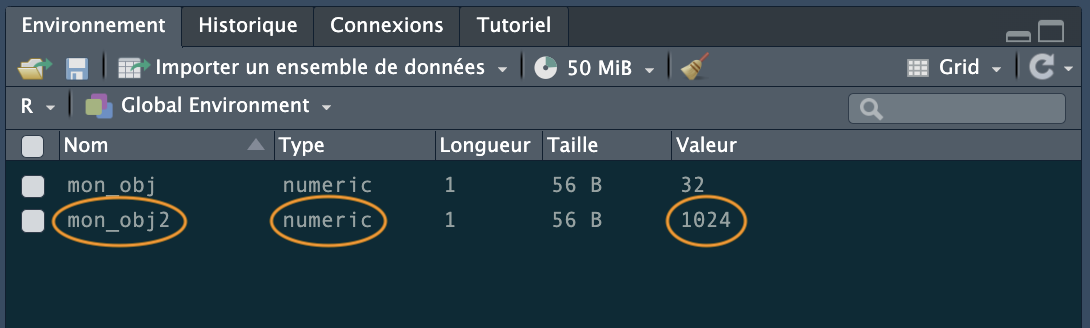
Une fois que nous avons créé plusieurs objets, nous pouvons faire des choses avec. Par exemple, le code suivant crée un nouvel objet mon_obj3 et lui assigne la valeur de mon_obj ajouté à mon_obj2 soit 1056 (32 + 1024 = 1056).
mon_obj3 <- mon_obj + mon_obj2
mon_obj3[1] 1056Remarquez que pour afficher la valeur de mon_obj3 nous devons également écrire le nom de l’objet. Le code ci-dessus fonctionne parce que les valeurs de mon_obj et mon_obj2 sont numériques (donc des nombres). Si vous essayez de faire ça avec des objets dont les valeurs sont des caractères (classe character), vous recevrez une erreur
char_obj <- "hello"
char_obj2 <- "world!"
char_obj3 <- char_obj + char_obj2
# Error in char_obj+char_obj2:non-numeric argument to binary operatorLe message d’erreur vous indique que l’un ou les deux objets char_obj et char_obj2 n’est pas un nombre et ne peut donc pas être additionné.
Lorsque vous commencez à apprendre R, la gestion des erreurs et des avertissements peut être frustrante car ils sont souvent difficiles à comprendre (qu’est-ce qu’un argument ? qu’est-ce qu’un opérateur binaire ?). Une façon de trouver plus d’informations sur une erreur particulière est de rechercher une version généralisée du message d’erreur. Pour l’erreur ci-dessus, essayez de rechercher ‘non-numeric argument to binary operator error + r’ ou même ‘common r error messages’.
Un autre message d’erreur que vous obtiendrez assez souvent lorsque vous commencerez à utiliser R est Error : object 'XXX' not found (erreur : objet ‘XXX’ non trouvé). A titre d’exemple, regardez le code ci-dessous
mon_obj <- 48
mon_obj4 <- mon_obj + no_obj
# Error: object 'no_obj' not foundR renvoie un message d’erreur parce que nous n’avons pas encore créé (défini) l’objet no_obj. Un autre indice qu’il y a un problème avec ce code est que, si vous vérifiez votre environnement, vous verrez que l’objet mon_obj4 n’a pas été créé.
Nommer vos objets est l’une des choses les plus difficiles que vous ferez dans R. Idéalement, les noms de vos objets devraient être courts et informatifs, ce qui n’est pas toujours facile. Si vous devez créer des objets avec plusieurs mots dans leur nom, utilisez un trait de soulignement _ ou un point . entre les mots ou mettez les différents mots en majuscules. Nous préférons le format souligné _ et n’incluons jamais de majuscules dans les noms (appelé snake_case).
resume_sortie <- "mon analyse" # recommandé #
resume.sortie <- "mon analyse"
resumeSortie <- "mon analyse"Il y a également quelques limitations lorsqu’il s’agit de donner des noms aux objets. Un nom d’objet ne peut pas commencer par un chiffre ou un point suivi d’un chiffre (ex. 2ma_variable ou .2ma_variable). Vous devez également éviter d’utiliser des caractères non alphanumériques ou des accents dans vos noms d’objets (i.e. &, ^, /, !, é, è, etc). De plus, assurez-vous de ne pas nommer vos objets avec des mots réservés (i.e. TRUE, NA) et ce n’est jamais une bonne idée de donner à votre objet le même nom qu’une fonction intégrée. Une fonction qui revient plus souvent qu’on ne peut s’en souvenir est :
data <- read.table("monfichierdedonees", header = TRUE)Oui, data() est une fonction de R qui permet de charger ou de lister les ensembles de données disponibles dans les paquets.
Jusqu’à présent, nous avons créé des objets simples en assignant directement une valeur unique à un objet. Il est très probable que vous souhaitiez bientôt créer des objets plus compliqués au fur et à mesure que vous aurez de l’expérience sur R et que la complexité de vos tâches augmente. Heureusement, R dispose d’une multitude de fonctions pour vous aider à le faire. Vous pouvez considérer une fonction comme un objet qui contient une série d’instructions pour effectuer une tâche spécifique. L’installation de base de R est livrée avec de nombreuses fonctions déjà définies ou vous pouvez augmenter la puissance de R en installant l’un des 10 000 paquets actuellement disponibles. Une fois que vous aurez acquis un peu plus d’expérience dans l’utilisation de R, vous voudrez peut-être définir vos propres fonctions pour effectuer des tâches spécifiques à vos objectifs (plus d’informations à ce sujet dans Chapitre 5).
La première fonction que nous allons découvrir est la fonction c(). La fonction c() est l’abréviation de concaténer et nous l’utilisons pour joindre une série de valeurs et les stocker dans une structure de données appelée vecteur (plus d’informations sur les vecteurs dans Chapitre 3).
mon_vec <- c(2, 3, 1, 6, 4, 3, 3, 7)Dans le code ci-dessus, nous avons créé un objet appelé mon_vec et lui avons assigné une valeur en utilisant la fonction c(). Il y a quelques points très importants à noter ici. Premièrement, lorsque vous utilisez une fonction dans R, le nom de la fonction est toujours suivi d’une paire de parenthèses rondes (), même s’il n’y a rien entre les parenthèses. Deuxièmement, les arguments d’une fonction sont placés à l’intérieur des parenthèses rondes () et sont séparés par des virgules ,. Vous pouvez considérer un argument comme un moyen de personnaliser l’utilisation ou le comportement d’une fonction. Dans l’exemple ci-dessus, les arguments sont les nombres que nous voulons concaténer. Enfin, l’une des choses les plus délicates lorsque vous commencez à utiliser R est de savoir quelle fonction utiliser pour une tâche particulière et comment l’utiliser. Heureusement, chaque fonction est toujours associée à un document d’aide qui explique comment utiliser la fonction (plus d’informations à ce sujet plus tard Section 2.6) et une recherche rapide sur le web peut également vous aider.
Pour examiner la valeur de notre nouvel objet, nous pouvons simplement taper le nom de l’objet comme nous l’avons fait précédemment
mon_vec[1] 2 3 1 6 4 3 3 7Maintenant que nous avons créé un vecteur, nous pouvons utiliser d’autres fonctions pour faire des choses utiles avec cet objet. Par exemple, nous pouvons calculer la moyenne, la variance, l’écart-type et le nombre d’éléments de notre vecteur en utilisant les fonctions mean(), var(), sd() et length().
mean(mon_vec) # renvoie la moyenne de mon_vec[1] 3.625var(mon_vec) # renvoie la variance de mon_vec[1] 3.982143sd(mon_vec) # renvoie l'écart-type de mon_vec[1] 1.995531length(mon_vec) # renvoie le nombre d'éléments dnas mon_vec[1] 8Si nous voulons utiliser l’une de ces valeurs plus tard dans notre analyse, il nous suffit d’affecter la valeur obtenue à un autre objet.
moyenne_vec <- mean(mon_vec) # renvoie la moyenne de mon_vec
moyenne_vec[1] 3.625Il peut parfois être utile de créer un vecteur contenant une séquence régulière de valeurs par pas de un. Dans ce cas, nous pouvons utiliser un raccourci en utilisant le symbole :.
ma_seq <- 1:10 # créer une séquence régulière
ma_seq [1] 1 2 3 4 5 6 7 8 9 10ma_seq2 <- 10:1 # en ordre décroissant
ma_seq2 [1] 10 9 8 7 6 5 4 3 2 1D’autres fonctions utiles pour générer des vecteurs de séquences sont seq() et rep(). Par exemple, pour générer une séquence de 1 à 5 par pas de 0,5 :
ma_seq2 <- seq(from = 1, to = 5, by = 0.5)
ma_seq2[1] 1.0 1.5 2.0 2.5 3.0 3.5 4.0 4.5 5.0Ici, nous avons utilisé les arguments from = et to = pour définir les limites de la séquence et l’argument by = pour spécifier l’incrément (les pas) de la séquence. Jouez avec d’autres valeurs pour ces arguments afin de voir leur effet.
L’argument rep() vous permet de répliquer (répéter) des valeurs un certain nombre de fois. Pour répéter la valeur ‘2’, 10 fois :
ma_seq3 <- rep(2, times = 10) # répète '2', 10 fois
ma_seq3 [1] 2 2 2 2 2 2 2 2 2 2Vous pouvez également répéter des valeurs non numériques :
ma_seq4 <- rep("abc", times = 3) # répète ‘abc' 3 fois
ma_seq4[1] "abc" "abc" "abc"ou chaque élément d’une série :
ma_seq5 <- rep(1:5, times = 3) # répète la série de '1' à '5', 3 fois
ma_seq5 [1] 1 2 3 4 5 1 2 3 4 5 1 2 3 4 5ou des éléments d’une série :
ma_seq6 <- rep(1:5, each = 3) # répète chaque élément de la série 3 fois
ma_seq6 [1] 1 1 1 2 2 2 3 3 3 4 4 4 5 5 5On peut aussi répéter une série non séquentielle :
[1] 3 3 3 1 1 1 10 10 10 7 7 7Notez dans le code ci-dessus comment nous avons utilisé la fonction c() à l’intérieur de la fonction rep(). L’imbrication de fonctions nous permet de construire des commandes assez complexes à l’intérieur d’une seule ligne de code et est une pratique très courante dans l’utilisation de R. Cependant, il faut faire attention car trop de fonctions imbriquées peuvent rendre votre code difficile à comprendre pour les autres (et pour vous-même dans le futur !). Nous pourrions réécrire le code ci-dessus pour séparer explicitement les deux étapes de la génération de notre vecteur. L’une ou l’autre approche donnera le même résultat, il vous suffit d’utiliser votre propre jugement pour déterminer laquelle est la plus lisible.
Manipuler, résumer et trier des données à l’aide de R est une compétence importante à maîtriser, mais que de nombreuses personnes trouvent un peu déroutante au début. Nous allons voir ici quelques exemples simples utilisant des vecteurs pour illustrer certains concepts importants, mais nous développerons cela plus en détail dans Chapitre 3 où nous verrons des structures de données plus compliquées (et plus utiles).
Pour extraire (ou indexer ou souscrire) une ou plusieurs valeurs (plus généralement appelées éléments) d’un vecteur, nous utilisons les crochets [ ]. L’approche générale consiste à nommer l’objet à extraire, puis écrire l’indice de l’élément à extraire dans les crochets. Cet indice peut être une position ou le résultat d’un test logique.
Pour extraire des éléments en fonction de leur position, il suffit d’écrire la position à l’intérieur des crochets [ ]. Par exemple, pour extraire la 3e valeur de mon_vec :
mon_vec # rappelons-nous à quoi mon_vec ressemble[1] 2 3 1 6 4 3 3 7mon_vec[3] # extrait la 3e valeur[1] 1# si vous voulez stocker cette valeur dans un autre objet
val_3 <- mon_vec[3]
val_3[1] 1Notez que l’indice de position commence à 1 et non à 0 comme dans d’autres langages de programmation (i.e. Python).
Nous pouvons également extraire plusieurs valeurs en utilisant la fonction c() à l’intérieur des crochets. Ici, nous extrayons le 1er, le 5e, le 6e et le 8e élément de l’objet mon_vec :
mon_vec[c(1, 5, 6, 8)][1] 2 4 3 7Nous pouvons également extraire une plage de valeurs à l’aide de la fonction :. Pour extraire du 3e au 8e élément :
mon_vec[3:8][1] 1 6 4 3 3 7Une autre façon très utile d’extraire des données d’un vecteur est d’utiliser une expression logique comme indice. Par exemple, pour extraire tous les éléments dont la valeur est supérieure à 4 dans le vecteur mon_vec :
mon_vec[mon_vec > 4][1] 6 7Ici, l’expression logique est mon_vec > 4 et R n’extraira que les éléments qui satisfont à cette condition logique. Comment cela fonctionne-t-il réellement ? Si nous regardons la sortie de l’expression logique sans les crochets, vous pouvez voir que R renvoie un vecteur contenant soit TRUE soit FALSE qui indique si la condition logique est remplie pour chaque élément. Dans ce cas, seuls les éléments en 4e et 8e position renvoient un TRUE car leur valeur est supérieure à 4.
mon_vec > 4[1] FALSE FALSE FALSE TRUE FALSE FALSE FALSE TRUEAinsi, ce que R fait en réalité sous le capot est équivalent à :
mon_vec[c(FALSE, FALSE, FALSE, TRUE, FALSE, FALSE, FALSE, TRUE)][1] 6 7et seuls les éléments qui sont TRUE seront extraits.
En plus de < et > vous pouvez également utiliser des opérateurs composites pour augmenter la complexité de vos expressions. Par exemple, l’expression “supérieur ou égal à” est : >=. Pour vérifier si une valeur est égale à une autre, nous devons utiliser un double symbole égal == et pour vérifier si une valeur est différente de, nous utilisons le symbole != (le symbole ! signifie “pas”).
mon_vec[mon_vec >= 4] # valeurs supérieures ou égales à 4[1] 6 4 7mon_vec[mon_vec < 4] # valeurs inférieures à 4[1] 2 3 1 3 3mon_vec[mon_vec <= 4] # valeurs inférieures ou égales à 4[1] 2 3 1 4 3 3mon_vec[mon_vec == 4] # valeurs égales à 4[1] 4mon_vec[mon_vec != 4] # valeurs pas égales à 4[1] 2 3 1 6 3 3 7Nous pouvons également combiner plusieurs expressions logiques à l’aide d’expressions booléennes. Dans R, l’élément & signifie ET et le symbole | signifie OU. Par exemple, pour extraire des valeurs dans mon_vec qui sont inférieures à 6 ET supérieures à 2 :
val26 <- mon_vec[mon_vec < 6 & mon_vec > 2]
val26[1] 3 4 3 3ou extraire des valeurs dans mon_vec qui sont supérieures à 6 OU inférieures à 3 :
val63 <- mon_vec[mon_vec > 6 | mon_vec < 3]
val63[1] 2 1 7Nous pouvons modifier les valeurs de certains éléments d’un vecteur à l’aide des crochets [ ] en combinaison avec l’opérateur d’affectation <-. Par exemple, pour remplacer la 4e valeur de mon_vec de 6 à 500 :
mon_vec[4] <- 500
mon_vec[1] 2 3 1 500 4 3 3 7Nous pouvons également remplacer plusieurs valeurs ou même remplacer des valeurs sur la base d’une expression logique :
# remplacer les 6e et 7e éléments par 100
mon_vec[c(6, 7)] <- 100
mon_vec[1] 2 3 1 500 4 100 100 7# remplacer les éléments inférieurs ou égaux à 4 par 1000
mon_vec[mon_vec <= 4] <- 1000
mon_vec[1] 1000 1000 1000 500 1000 100 100 7Outre l’extraction d’éléments particuliers d’un vecteur, il est également possible d’ordonner les valeurs contenues dans un vecteur. Pour trier les valeurs de la plus petite à la plus grande, nous pouvons utiliser la fonction sort() :
vec_trie <- sort(mon_vec)
vec_trie[1] 7 100 100 500 1000 1000 1000 1000Pour inverser le tri, du plus élevé au plus bas, nous pouvons soit inclure l’option decreasing = TRUE lors de l’utilisation de la fonction sort() :
vec_trie2 <- sort(mon_vec, decreasing = TRUE)
vec_trie2[1] 1000 1000 1000 1000 500 100 100 7soit trier d’abord le vecteur à l’aide de la fonction sort() puis l’inverser à l’aide de la fonction rev(). Il s’agit là d’un autre exemple d’imbrication d’une fonction dans une autre fonction :
Bien qu’il soit amusant de trier un seul vecteur, il serait peut-être plus utile de trier un vecteur en fonction des valeurs d’un autre vecteur. Pour ce faire, nous devons utiliser la fonction order() en combinaison avec [ ]. Pour le démontrer, créons un vecteur appelé taille contenant la taille de 5 personnes différentes et un autre vecteur appelé p_noms contenant les noms de ces personnes (Joanna mesure 180 cm, Charlotte mesure 155 cm, etc.)
taille <- c(180, 155, 160, 167, 181)
taille[1] 180 155 160 167 181p_noms <- c("Joanna", "Charlotte", "Helen", "Karen", "Amy")
p_noms[1] "Joanna" "Charlotte" "Helen" "Karen" "Amy" Notre objectif est de classer les personnes dans p_noms dans l’ordre croissant de leur taille. La première chose que nous allons faire est d’utiliser la fonction order() avec le vecteur taille pour créer un vecteur appelé taille_ord
taille_ord <- order(taille)
taille_ord[1] 2 3 4 1 5OK, que se passe-t-il ici ? La première valeur, 2(n’oubliez pas d’ignorer [1]) doit être lue comme “la plus petite valeur de taille est le deuxième élément du vecteur taille”. Si nous le vérifions en regardant le vecteur taille ci-dessus, nous pouvons voir que le 2e élément a une valeur de 155, ce qui est la plus petite valeur. La deuxième valeur la plus petite du vecteur taille est la 3e ce qui, après vérification, donne 160 et ainsi de suite. La plus grande valeur de taille est la 5e qui vaut 181. Maintenant que nous avons le vecteur des indices de position des tailles par ordre croissant (taille_ord), nous pouvons extraire ces valeurs de notre vecteur p_noms dans cet ordre
noms_ord <- p_noms[taille_ord]
noms_ord[1] "Charlotte" "Helen" "Karen" "Joanna" "Amy" Vous vous demandez probablement à quoi cela peut bien servir. Imaginons que vous disposiez d’un jeu de données contenant deux colonnes et que vous souhaitiez trier chacune d’entre elles. Si vous utilisez simplement sort() pour trier chaque colonne séparément, les valeurs de chaque colonne seront dissociées les unes des autres. En utilisant order() sur une colonne, un vecteur d’indices de position est créé à partir des valeurs de la colonne dans l’ordre croissant. Ce vecteur peut être utilisé sur la deuxième colonne, en tant qu’indice d’éléments qui renverront un vecteur de valeurs basé sur la première colonne. En toute honnêteté, lorsque vous avez plusieurs vecteurs liés, vous devez utiliser un objet de type data.frame (voir Chapitre 3) au lieu de plusieurs vecteurs indépendants.
L’un des avantages des fonctions R est que la plupart d’entre elles sont vectorisées. Cela signifie que la fonction opère sur tous les éléments d’un vecteur sans qu’il soit nécessaire d’appliquer la fonction à chaque élément séparément. Par exemple, pour multiplier chaque élément d’un vecteur par 5, il suffit d’utiliser la fonction :
# créer un vecteur
mon_vec2 <- c(3, 5, 7, 1, 9, 20)
# multiplier chaque élément par 5
mon_vec2 * 5[1] 15 25 35 5 45 100Ou nous pouvons additionner les éléments de deux vecteurs ou plus :
# créer un deuxième vecteur
mon_vec3 <- c(17, 15, 13, 19, 11, 0)
# additionner les 2 vecteurs
mon_vec2 + mon_vec3[1] 20 20 20 20 20 20# multiplier les 2 vecteurs
mon_vec2 * mon_vec3[1] 51 75 91 19 99 0Cependant, vous devez faire attention lorsque vous utilisez la vectorisation avec des vecteurs de longueurs différentes, car R recyclera tranquillement les éléments du vecteur le plus court plutôt que de signaler une erreur.
# créer un troisième vecteur
mon_vec4 <- c(1, 2)
# additionner les 2 vecteurs - recyclage tranquille!
mon_vec2 + mon_vec4[1] 4 7 8 3 10 22Dans R, les données manquantes sont généralement représentées par un NA qui signifie “Not Available” (Non disponible). Les données peuvent être manquantes pour toute une série de raisons : votre machine est peut-être tombée en panne, vous êtes peut-être tombé en panne, le temps était peut-être trop mauvais pour collecter des données un jour donné, etc. Les données manquantes peuvent être une véritable plaie, tant du point de vue de R que du point de vue statistique. Du point de vue de R, les données manquantes peuvent être problématiques car différentes fonctions traitent les données manquantes de différentes manières. Par exemple, supposons que nous ayons recueilli des relevés de température de l’air pendant 10 jours, mais que notre thermomètre se soit cassé le deuxième et le neuvième jour, de sorte que nous n’avons pas de données pour ces jours-là :
temp <- c(7.2, NA, 7.1, 6.9, 6.5, 5.8, 5.8, 5.5, NA, 5.5)
temp [1] 7.2 NA 7.1 6.9 6.5 5.8 5.8 5.5 NA 5.5Nous voulons maintenant calculer la température moyenne sur ces jours à l’aide de la fonction mean() :
temp_moyenne <- mean(temp)
temp_moyenne[1] NASi un vecteur a une valeur manquante, la seule valeur possible à renvoyer lors du calcul d’une moyenne est NA. R ne sait pas que vous souhaitez peut-être ignorer la valeur NA (R ne peut pas lire dans vos pensées - pour l’instant !). Si nous regardons le fichier d’aide (en utilisant ?mean - voir la section suivante Section 2.6 pour plus de détails) associé à la fonction mean() nous pouvons voir qu’il y a un argument na.rm = qui prend la valeur FALSE par défaut.
na.rm - une valeur logique indiquant si les valeurs NA doivent être supprimées avant le calcul (“na remove”).
Si nous remplaçons cet argument par na.rm = TRUE lorsque nous utilisons la fonction mean() cela nous permettra d’ignorer les NA lors du calcul de la moyenne :
temp_moyenne <- mean(temp, na.rm = TRUE)
temp_moyenne[1] 6.2875Il est important de noter que les NA n’ont pas été retirés du vecteur temp (ce serait une mauvaise pratique), mais que l’objet mean() les a simplement ignorées. Le but de ce qui précède est de souligner comment nous pouvons modifier le comportement par défaut d’une fonction à l’aide d’un argument approprié. Le problème est que toutes les fonctions n’ont pas d’argument na.rm = elles peuvent gérer les NA différemment. Cependant, la bonne nouvelle est que chaque fichier d’aide associé à une fonction vous indiquera toujours comment les données manquantes sont traitées par défaut.
Ce livre est conçu comme une introduction relativement brève à R et, en tant que tel, vous utiliserez bientôt des fonctions et des paquets qui dépassent le cadre de ce texte d’introduction. Heureusement, l’une des forces de R est son système d’aide complet et facilement accessible, ainsi que la richesse des ressources en ligne où vous pouvez obtenir de plus amples informations.
Pour accéder à l’aide intégrée de R et obtenir des informations sur n’importe quelle fonction, il suffit d’utiliser la fonction help(). Par exemple, pour ouvrir la page d’aide de notre amie, la fonction mean() :
help("mean")ou vous pouvez utiliser le raccourci ? devant la fonction :
?meanla page d’aide est affichée dans l’onglet “Aide”(généralement en bas à droite sur RStudio)
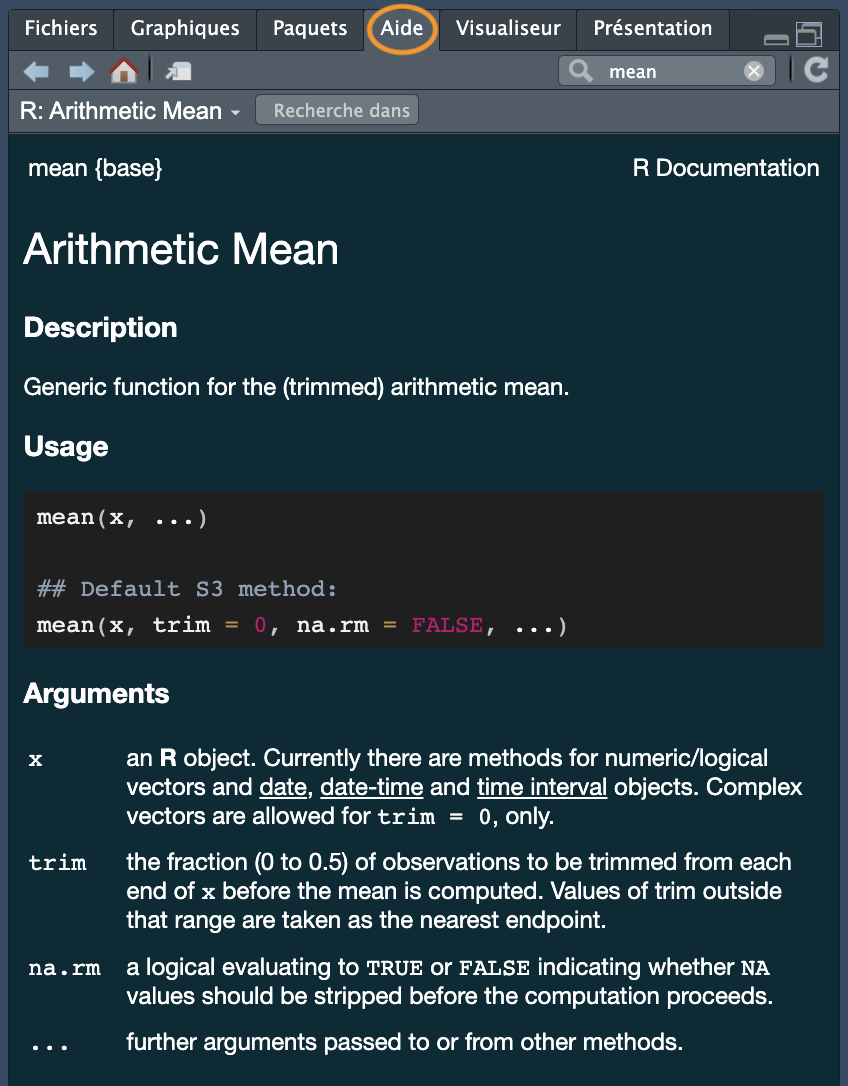
mean() dans le panneau Aide sur Rstudio
Il est vrai que les fichiers d’aide peuvent sembler tout sauf utiles lorsque vous commencez à utiliser R. Cela est probablement dû au fait qu’ils sont écrits de manière très concise et que le langage utilisé est souvent assez technique et plein de jargon. Cela dit, on s’y habitue et, avec le temps, on finit même par apprécier une certaine beauté dans cette brièveté (honnêtement !). L’un des aspects les plus intéressants des fichiers d’aide est qu’ils ont tous une structure très similaire, quelle que soit la fonction. Il est donc facile de naviguer dans le fichier pour trouver exactement ce dont vous avez besoin.
La première ligne du document d’aide contient des informations telles que le nom de la fonction et le paquet d’où elle provient (entre les accolades {}, ici {base} signifie que la fonction mean() fait partie des fonctions de base de R). D’autres rubriques fournissent des informations plus spécifiques, telles que
| Rubriques | Description de la rubrique |
|---|---|
| Description : | donne une brève description de la fonction et de ce qu’elle fait. |
| Usage : | donne le nom des arguments associés à la fonction et les éventuelles valeurs par défaut. |
| Arguments : | fournit plus de détails sur chaque argument et sur ce qu’il fait. |
| Details : | donne des détails supplémentaires sur la fonction si nécessaire. |
| Value : | le cas échéant, indique le type et la structure de l’objet renvoyé par la fonction ou l’opérateur. |
| See also : | fournit des informations sur d’autres pages d’aide au contenu similaire ou connexe. |
| Examples : | donne quelques exemples d’utilisation de la fonction. |
Les Examples (Exemples d’application) sont très utiles, il suffit de les copier et de les coller dans la console pour voir ce qui se passe. Vous pouvez également accéder aux exemples à tout moment en utilisant la fonction example() (c’est-à-dire example("mean"))
La fonction help() est utile si vous connaissez le nom de la fonction. Si vous n’êtes pas sûr du nom, mais que vous vous souvenez d’un mot clé, vous pouvez faire une recherche dans le système d’aide de R à l’aide de la fonction help.search().
help.search("mean")ou vous pouvez utiliser le raccourci équivalent ?? :
??meanLes résultats de la recherche seront affichés dans RStudio sous l’onglet “Aide” comme précédemment. help.search() recherche dans la documentation d’aide, les démonstrations de code et les vignettes de paquet et affiche les résultats sous forme de liens cliquables pour une exploration plus approfondie.
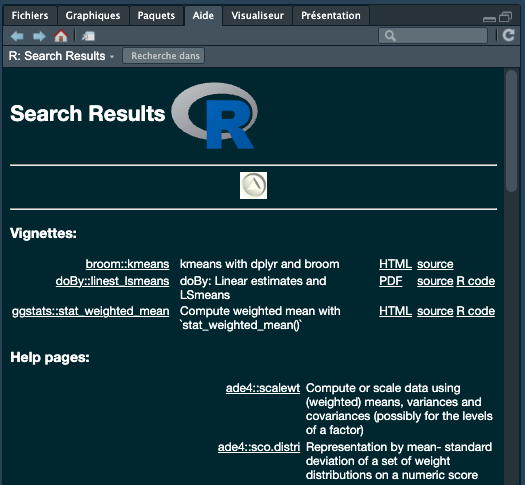
help.search() dans Rstudio
Une autre fonction utile est apropos(). Cette fonction peut être utilisée pour dresser la liste de toutes les fonctions contenant une chaîne de caractères spécifiée. Par exemple, pour trouver toutes les fonctions avec mean dans leur nom :
apropos("mean") [1] ".colMeans" ".rowMeans" "colMeans" "kmeans"
[5] "mean" "mean_temp" "mean.Date" "mean.default"
[9] "mean.difftime" "mean.POSIXct" "mean.POSIXlt" "rowMeans"
[13] "vec_mean" "weighted.mean"Vous pouvez alors afficher le fichier d’aide de la fonction concernée.
help("kmeans")Une autre fonction est RSiteSearch() qui vous permet de rechercher des mots-clés et des phrases dans les pages d’aide des fonctions et les vignettes de tous les paquets CRAN. Cette fonction vous permet d’accéder au moteur de recherche du site web de R https://www.r-project.org/search.html directement à partir de la console et d’afficher les résultats dans votre navigateur web.
RSiteSearch("regression")Il n’y a jamais eu de meilleur moment pour commencer à apprendre R. Il existe pléthore de ressources en ligne disponibles gratuitement, allant de cours complets à des tutoriels et des listes de diffusion spécifiques à un sujet. Il existe également de nombreuses options payantes si c’est votre truc, mais à moins que vous n’ayez de l’argent à brûler, il n’est vraiment pas nécessaire de dépenser votre argent durement gagné. Vous trouverez ci-dessous quelques ressources que nous avons trouvées utiles.
Votre approche de l’enregistrement du travail dans R et RStudio dépend de ce que vous voulez enregistrer. La plupart du temps, la seule chose que vous devrez sauvegarder est le code R de vos scripts. N’oubliez pas que votre script est un enregistrement reproductible de tout ce que vous avez fait. Il vous suffit donc d’ouvrir votre script dans une nouvelle session RStudio et de l’exécuter dans la console R pour revenir à l’endroit où vous vous étiez arrêté.
À moins que vous n’ayez suivi notre suggestion de modifier les paramètres par défaut des projets RStudio (voir Section 1.5), il vous sera demandé si vous souhaitez sauvegarder l’image de votre espace de travail à chaque fois que vous quitterez RStudio. Nous pensons que dans 99,9 % des cas, vous ne souhaitez pas le faire. En commençant avec une session RStudio propre chaque fois que nous revenons à notre analyse, nous pouvons être sûrs d’éviter tout conflit potentiel avec les choses que nous avons faites dans les sessions précédentes.
Cependant, il est parfois utile de sauvegarder les objets que vous avez créés dans R. Par exemple, imaginons que vous créiez un objet dont la génération nécessite des heures (voire des jours) de temps de calcul. Il serait extrêmement gênant de devoir attendre tout ce temps à chaque fois que vous revenez sur votre analyse. Cependant, dans ce cas, nous pouvons enregistrer cet objet en tant que fichier externe, .RData que nous pourrons charger dans RStudio la prochaine fois que nous voudrons l’utiliser. Pour enregistrer un objet dans un fichier .RData vous pouvez utiliser la fonction save() (remarquez que nous n’avons pas besoin d’utiliser l’opérateur d’affectation ici) :
save(nomDelObjet, file = "nom_du_fichier.RData")ou si vous souhaitez sauvegarder tous les objets de votre espace de travail dans un seul fichier .RData utilisez la fonction save.image() :
save.image(file = "nom_du_fichier.RData")Pour charger votre .RData dans RStudio, utilisez la fonction load() :
load(file = "nom_du_fichier.RData")L’installation de base de R est livrée avec de nombreux paquets utiles. Ces paquets contiennent de nombreuses fonctions que vous utiliserez quotidiennement. Cependant, lorsque vous commencerez à utiliser R pour des projets plus variés (et que votre propre utilisation de R évoluera), vous constaterez qu’il y a un moment où vous aurez besoin d’étendre les capacités de R. Heureusement, des milliers d’utilisateurs de R ont développé du code utile et l’ont partagé sous forme de paquets installables. Vous pouvez considérer un paquet comme une collection de fonctions, de données et de fichiers d’aide rassemblés dans une structure standard bien définie que vous pouvez télécharger et installer dans R. Ces paquets peuvent être téléchargés à partir de diverses sources, mais les plus populaires sont les suivantes CRAN, Bioconductor et GitHub . Actuellement, le CRAN héberge plus de 15 000 paquets et est le dépôt officiel des paquets R fournis par les utilisateurs. Bioconductor fournit des logiciels libres orientés vers la bioinformatique et héberge plus de 1800 paquets R. GitHub est un site web qui héberge des dépôts git pour toutes sortes de logiciels et de projets (pas seulement R). Souvent, les versions de développement de pointe des paquets R sont hébergées sur GitHub, donc si vous avez besoin de toutes les nouvelles fonctionnalités, cela peut être une option. Cependant, l’inconvénient potentiel de l’utilisation de la version de développement d’un paquet R est qu’elle peut ne pas être aussi stable que la version hébergée sur CRAN (elle est en cours de développement !) et que la mise à jour des paquets ne sera pas automatique.
Une fois que vous avez installé un paquet sur votre ordinateur, vous ne pouvez pas l’utiliser immédiatement. Pour utiliser un paquet, vous devez d’abord le charger à l’aide de la fonction library(). Par exemple, pour charger le paquet remotes 📦 que vous avez installé précédemment :
La fonction library() chargera également tous les paquets supplémentaires nécessaires et pourra afficher des informations supplémentaires sur les paquets dans la console. Il est important de savoir que chaque fois que vous démarrez une nouvelle session R (ou que vous restaurez une session précédemment sauvegardée), vous devez charger les paquets que vous utiliserez. Nous avons tendance à mettre tous nos library() nécessaires à notre analyse en tête de nos scripts R afin de les rendre facilement accessibles et de pouvoir les compléter au fur et à mesure du développement de notre code. Si vous essayez d’utiliser une fonction sans avoir préalablement chargé le paquet R correspondant, vous recevrez un message d’erreur indiquant que R n’a pas pu trouver la fonction. Par exemple, si vous essayez d’utiliser la fonction install_github() sans charger le paquet remotes 📦 en premier lieu, vous obtiendrez l’erreur suivante :
install_github("tidyverse/dplyr")
# Error in install_github("tidyverse/dplyr") :
# could not find function "install_github"Il peut parfois être utile d’utiliser une fonction sans utiliser au préalable la fonctionlibrary(). Si, par exemple, vous n’utilisez qu’une ou deux fonctions dans votre script et que vous ne souhaitez pas charger toutes les autres fonctions d’un paquet, vous pouvez accéder directement à la fonction en spécifiant le nom du paquet, suivi de deux points (2 fois) ::, puis du nom de la fonction :
remotes::install_github("tidyverse/dplyr")C’est ainsi que nous avons pu utiliser la fonction install() et install_github() ci-dessous sans charger les paquets au préalableBiocManager 📦 et remotes 📦 . La plupart du temps, nous recommandons d’utiliser la fonction library().
Pour installer un paquet à partir du CRAN, vous pouvez utiliser la fonction install.packages(). Par exemple, si vous voulez installer le paquet remotes 📦 entrez le code suivant dans la Console (note : vous aurez besoin d’une connexion internet fonctionnelle pour effectuer cette opération) :
install.packages("remotes", dependencies = TRUE)Il vous sera peut-être demandé de choisir un miroir CRAN, sélectionnez simplement ‘0-cloud’ ou un miroir proche de votre localisation. L’argument dependencies = TRUE permet de s’assurer que les paquets supplémentaires nécessaires seront également installés.
Il est conseillé de mettre régulièrement à jour les paquets déjà installés afin de bénéficier des nouvelles fonctionnalités et des corrections de bogues. Pour mettre à jour les paquets CRAN, vous pouvez utiliser la commande update.packages() (vous aurez besoin d’une connexion internet pour cela) :
update.packages(ask = FALSE)L’argument ask = FALSE évite d’avoir à confirmer chaque téléchargement de paquet, ce qui peut être fastidieux si de nombreux paquets sont installés.
Pour installer des paquets de Bioconductor, le processus est un peu différent. Vous devez d’abord installer le paquet BiocManager 📦. Vous ne devez le faire qu’une seule fois, sauf si vous réinstallez ou mettez à jour R.
install.packages("BiocManager", dependencies = TRUE)Une fois que BiocManager a été installé, vous pouvez soit installer tous les paquets “de base” de Bioconductor avec la commande :
BiocManager::install()ou installer des paquets spécifiques tels que le GenomicRanges 📦 et edgeR 📦 :
BiocManager::install(c("GenomicRanges", "edgeR"))Pour mettre à jour les paquets de Bioconductor, il suffit d’utiliser la commande BiocManager::install() à nouveau :
BiocManager::install(ask = FALSE)Là encore, vous pouvez utiliser l’argument ask = FALSE pour éviter d’avoir à confirmer chaque téléchargement de paquet.
Il existe plusieurs options pour installer les paquets hébergés sur GitHub. La méthode la plus efficace est sans doute d’utiliser la fonction install_github() du paquet remotes 📦 (vous avez installé ce paquet précédemment, Section 2.8.2.1). Avant d’utiliser la fonction, vous devez connaître le nom d’utilisateur GitHub du propriétaire du répertoire ainsi que le nom du répertoire. Par exemple, la version de développement de dplyr 📦 de Hadley Wickham est hébergée sur le compte GitHub de tidyverse et porte le nom de répertoire “dplyr” (recherchez simplement “github dplyr”). Pour installer cette version depuis GitHub, utilisez :
remotes::install_github("tidyverse/dplyr")Le moyen le plus sûr (à notre connaissance) de mettre à jour un paquet installé depuis GitHub est de le réinstaller en utilisant la commande ci-dessus.